製品・サービス情報
|
|
ArrayStar社 遺伝子発現制御機構解析
non-coding RNA マイクロアレイ受託解析サービス |
|
 |
| Arraystar社の各種アレイを用いた受託解析サービスをご提供いたします。ベストセラーであるLncRNAマイクロアレイをはじめ、CircRNAマイクロアレイ,
T-UCRマイクロアレイ, piRNAマイクロアレイの受託サービスを提供しています。 |
 LncRNA Arrays 受託解析サービス LncRNA Arrays 受託解析サービス

Long non-coding RNA(LncRNA)は、タンパク質をコードしない200塩基長のRNAであり、生物学的プロセスやヒトの疾患に関与しています。
Arraystar社の LncRNA Arraysは、LncRNAおよびタンパク質コードmRNAを体系的にプロファイリングする様デザインされています。このアレイは、包括的かつ厳密に収集された最高のLncRNAコンテンツ(Reliable
LncRNA)を有し、十分にアノテーションされ、また、実験的に裏付けされたLncRNAのデータソースも利用されます(Gold Standard
LncRNA)。パフォーマンスの高いワークフローにより、RNA-Seqよりも優れたLncRNAプロファイリングデータを提供します。専用のLncRNAデータ解析およびアノテーションは、複雑なLncRNA生物学の解明やタンパク質コード遺伝子との関連性を明らかにする際に、重要な役割を果たしています。
【特長】
|
◆LncRNAおよびmRNAのプロファイリング
◆Transcript-specificプローブの採用による正確な転写産物アイソフォームの検出
◆質の高いLncRNAデータベース(Reliable LncRNA, Gold Standard LncRNA) |
|
|

カタログダウンロード(PDF) |
|
◆RNA-Seqよりも優れたLncRNA発現のプロファイリング
|
マイクロアレイ技術は、これまで遺伝子発現の基本プラットフォームとして利用されてきましたが、近年では、次世代シークエンシング(NGS)が遺伝子発現研究において頻用される様になっています。NGSでは、全トランスクリプトームにおける転写産物のシークエンスリードを定量することによって、遺伝子発現レベルを測定します(RNA-Seq)。しかしながら、LncRNA発現において、マイクロアレイ技術はRNA-Seqと比較して、重要かつ代替することのできない利点を有しています。
⇒詳しくはこちら
・LncRNAは、タンパク質コードRNAと比較して発現レベルが低いため、RNA-Seqでは、低含有転写産物の信頼性のある定量が困難。
・RNA-Seqでは、リード深度の増強による低含有量転写産物の正確性に対する効果は限定的
・マイクロアレイは、LncRNAを包括的に網羅し、低含量LncRNAもプロファイリング
・マイクロアレイは、充分に確立され、かつ成熟した技術 |
◆質の高いLncRNAデータベース(Reliable LncRNA, Gold Standard LncRNA
|
タンパク質コード遺伝子と異なり、公的に利用可能なLncRNAデータベースではアノテーションが不充分な場合があります。これに対し、本サービスでは、高品質なトランスクリプトームおよび
LncRNAデータベースであるGold Standard LncRNA および Reliable LncRNA を有しています。これらのデータベースは、47Tb以上のRNA-Seqデータや主要な公的データベース(Refseq,
USCS known Genes, GENCODE, lncRNA catalogs, lncRNAdb, T-UCRs, RNAdb, NRED,
および文献)に基づいています。
⇒詳しくはこちら |
|
【データベース】
|
◆Human LncRNA Expression Array V5.0
|
| プローブ総数 |
60,491 |
| プローブ長 |
60 nt |
| プローブ選択領域 |
mRNAとLncRNA:転写産物の全長に沿った特定のエクソンまたはスプライスジャンクション |
| プローブ特異性 |
転写物特異的 |
| 総LncRNA |
39,917(内8,393のGold Standard LncRNAと30,924のReliable LncRNA) |
| タンパク質コード mRNA |
21,174 |
| LncRNAソース |
Arraystar LncRNA コレクションパイプライン:
2018年までのすべての主要なデータベースおよび文献からのLncRNA。各lncRNA遺伝子の転写物に優先的に「Canonical」または「longest」を割り当て。
外部データベース(2018年現在):
FANTOM5 CAT(v1)、GENECODE(v29)、RefSeq(Updated to 2018.11)、BIGTranscriptome(v1)、KnownGene(Updated
to 2018.11)、LncRNAdb、LncRNAWiki、RNAdb、NRED、CLS FL、NONCODE(v5)、MiTranscriptome(v2)
文献: 2018年までの科学出版物 |
| mRNAソース |
RefSeq、UCSC、GENECODE、FANTOM5 CAT |
| アレイフォーマット |
8×60K |
|
◆Mouse LncRNA Expression Array V4.0
|
| プローブ総数 |
60,641 |
| プローブ長 |
60 nt |
| プローブ選択領域 |
mRNAとLncRNA:転写産物の全長に沿った特定のエクソンまたはスプライスジャンクション |
| プローブ特異性 |
転写物特異的 |
| 総LncRNA |
37,949 |
| タンパク質コード mRNA |
22,692 |
| LncRNAソース |
Arraystar LncRNA コレクションパイプライン:
2018年までのすべての主要なデータベースおよび文献からのLncRNA。各lncRNA遺伝子の転写物に優先的に「Canonical」または「longest」を割り当て。
外部データベース(2018年現在):
GENECODE(v19)、RefSeq、KnownGene、GenBank
文献: 2018年までの科学出版物 |
| mRNAソース |
RefSeq、Known Gene、GENECODE |
| アレイフォーマット |
8×60K |
|
◆Rat LncRNA Expression Microarray V3.0
|
| プローブ総数 |
38,352 |
| プローブ長 |
60 nt |
| プローブ選択領域 |
mRNAとLncRNA:転写産物の全長に沿った特定のエクソンまたはスプライスジャンクション |
| プローブ特異性 |
転写物特異的 |
| 総LncRNA |
10,333 |
| タンパク質コード mRNA |
28,287 |
| LncRNAソース |
Arraystar LncRNA コレクションパイプライン:
2018年までのすべての主要なデータベースおよび文献からのLncRNA。各lncRNA遺伝子の転写物に優先的に「Canonical」または「longest」を割り当て。
外部データベース(2018年現在):
RefSeq、Ensembl
文献: 2018年までの科学出版物 |
| mRNAソース |
RefSeq、Ensembl |
| アレイフォーマット |
4×44K |
|
|
【ワークフロー】
|
本サービスでは、サンプル調整からデータ解析まで、LncRNAマイクロアレイのプロファイリングに関する全てのサービスを提供します。各ステップにおける品質管理は、信頼できる結果の取得を確かなものとする様にデザインされています。お客様にはサンプルをご提供頂くだけです。
サンプルの準備については、Sample Submission Guideline をご参照ください。
・RNA 品質チェック(QC)
・cDNA 合成
・Cy3 標識によるターゲット調整
・アレイハイブリダイゼーション、洗浄、スキャニング
・データ抽出、解析、要約 |
【バイオインフォマティクス解析の概要とその一例】
|
◆解析概要
|
| LncRNA |
mRNA |
| ・ |
Rawデータの正規化および低シグナルデータのフィルタリング |
| ・ |
Gold Standard LncRNAおよびReliable LncRNAによるアノテーション |
| ・ |
発現変動LncRNAスクリーニング |
| ・ |
ヒートマップおよびクラスター分析 |
|
| ・ |
Rawデータの正規化および低シグナルデータのフィルタリング |
| ・ |
発現変動mRNAのスクリーニング |
| ・ |
ヒートマップおよびクラスター解析 |
| ・ |
パスウェイ解析 |
| ・ |
Gene Ontology(GO)解析 |
|
| LncRNA分類およびサブグループ解析, Gold Standard LncRNA と Reliable LncRNA のアノテーション |
| ・ |
発現変動LncRNAおよびその隣接コード遺伝子 |
| ・ |
発現変動antisense LncRNA およびその隣接コード遺伝子 |
|
|
◆データ例
|
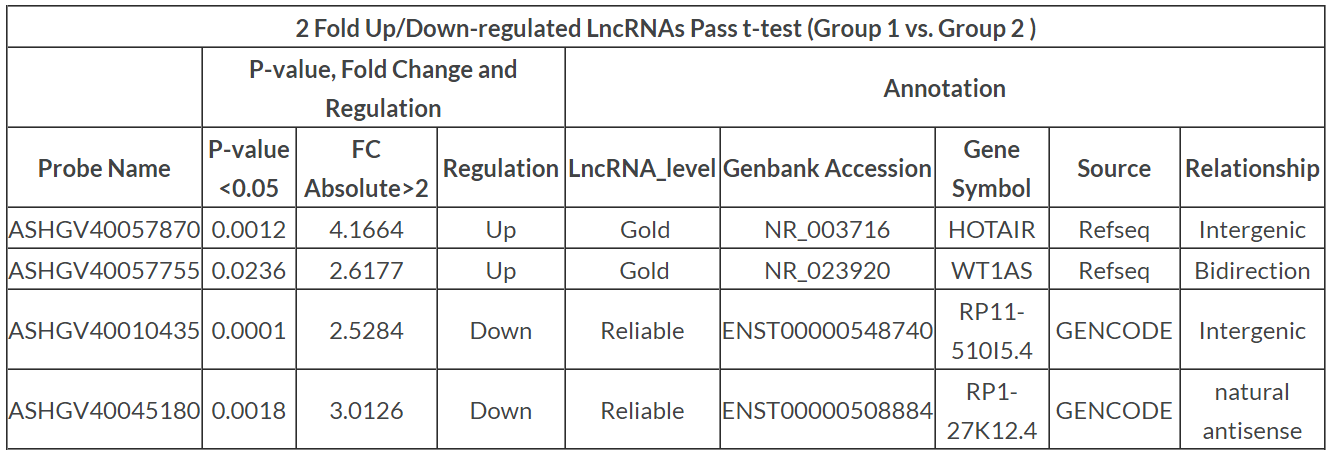
表1. 発現変動したLncRNAのスクリーニング。グループ1と2における発現変動LncRNA(Fold change>2, p-value<0.05)。
「Regulation」列の「Up」はアップレギュレーションを、「Down」はダウンレギュレーションを表す。
 |
|
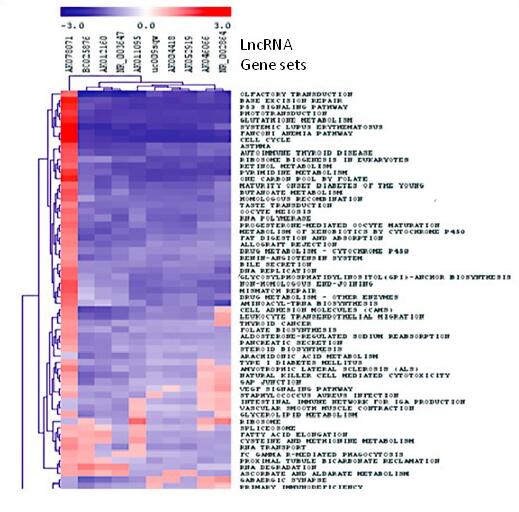 |
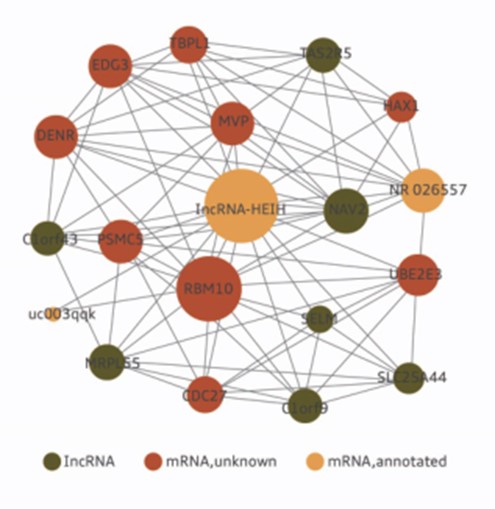
図1.
肝細胞がんにおけるLncRNA-HEIHのコード・非コード相互作用共発現(CNC)サブネットワーク |
|
図2.
生物学的機能におけるlncRNAのエンリッチメントを同定するためのLnc-GSET |
|
|
【参考文献】
【税別価格】
|
| アレイ名 |
生物種 |
サンプル数 |
税別単価※ |
Cat.# |
| LncRNA Array 受託解析サービス |
ヒト |
1サンプルから受付 |
¥175,000 |
F-AS-LNCH-(サンプル数) |
| マウス |
¥175,000 |
F-AS-LNCM-(サンプル数) |
| ラット |
¥155,000 |
F-AS-LNCR-(サンプル数) |
※1サンプルの解析料金です。海外提携先への送料が別途必要となります。 |
|
|
 解析の流れと必要サンプル量 解析の流れと必要サンプル量

*本サイトの情報は、Arraystar社のHomepageの情報を一部引用しております。
|
|