製品・サービス情報
|
|
BioTuring社製品 シングルセル RNA-Seq 解析ソフトウェア
BBrowserX Pro |
|
|
 |
 BBrowserX Pro BBrowserX Pro

| |
【個人データを使用したシングルセル RNA-Seq データ解析】
| |
BBrowserX Pro は、シングルセル RNA-Seq データ解析のエンドツーエンドのソリューションです。様々なデータのインポート・グラフ化・下流分析に対応しています。計算・データ保存は、メーカーのクラウド上で行われます。
|
|
|
 |
| |
| |
コンピュータープログラミングの知識がないウェットラボの研究者でも、自身のシングルセルデータを分析し、解析ワークフローを直感的に理解できる様に設計されています。データの可視化から解析までを一貫してサポートし、得られたデータから有用な洞察を引き出すことができます。
また、Scanpy パイプラインを基盤とし、Seurat の各種パッケージを補完する BBrowserX Pro は、すでにシングルセル RNA-seq
解析に精通している研究者にも最適なプラットフォームです。 |
|
 特長 特長

| |
◆様々なファイル形式に対応
| |
BBrowserX Pro は、数百万セル以上の大規模データセットのアップロードをサポートしています。以下のファイル形式がサポートされています。
AnnData object (.h5ad)
Seurat (.rds)
10x Genomics Cell Ranger (マトリクス)
10x Genomics Cell Ranger (.h5)
Data table (.tsv/.csv)
MuData object (.h5mu)
TileDB Soma
また、BBrowserX Pro では、解析がパイプライン化されているため、データをインポートするだけで UMAP が自動生成されます。 |
|
|
 |
◆細胞種を自動で予測
| |
セルクラスターがどの様な細胞種であるかをラベリングする「アノテーション」は、シングルセル RNA-seq 解析において重要なプロセスであると同時に、時間を要する解析の一つです。
BioTurning 社は、世界最大級の厳選されたシングルセルデータベースを構築しており、このデータベースを活用したマーカー遺伝子ベースの自動細胞種アノテーションをサポートしています。加えて、各クラスターについて主要なマーカー遺伝子や研究概要、論文リンク、著者による細胞種アノテーション情報を確認できるため、最終的なアノテーションの妥当性を慎重に評価することができます。
さらに、独自のデータセットから自動でアノテーションを付与することも可能です。 |
|
|
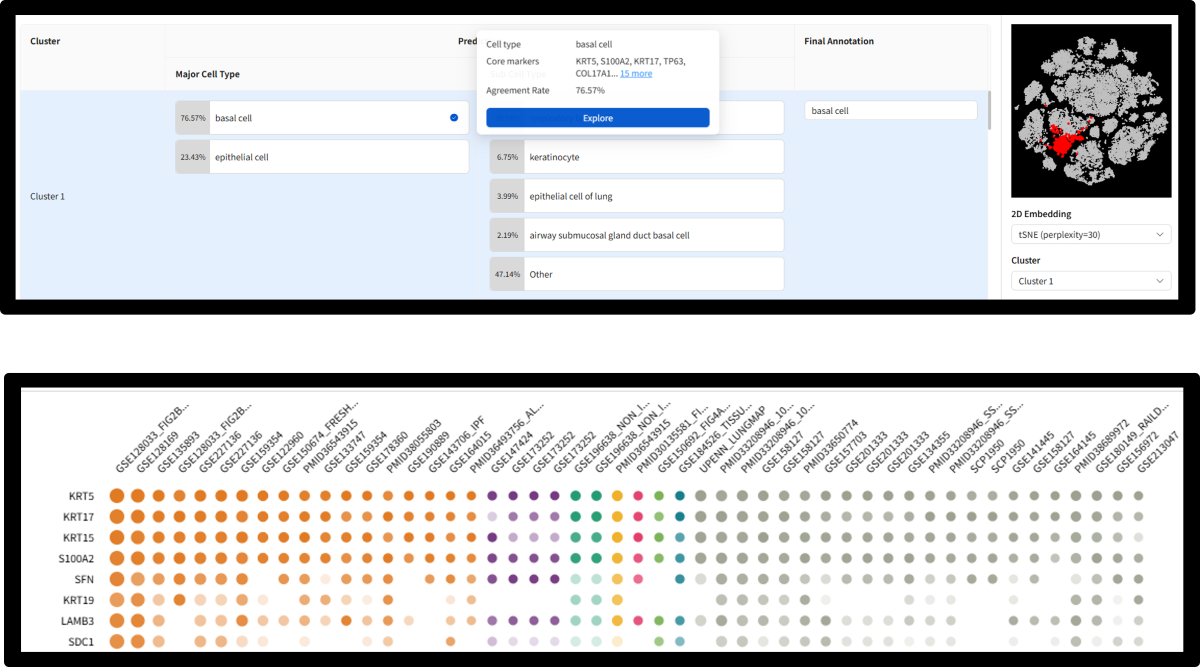
自動細胞種アノテーション
(クリックして拡大) |
◆がん細胞の予測
| |
シングルセル RNA-seq データセットでは、正常細胞とがん細胞が混在することが予想されます。従って、腫瘍の生物学的性質を理解するには、これらの
2 種類の細胞を区別する必要があります。
BBrowserX Pro は、コピー数変異(CNV)に基づいてがん細胞を予測します。inferCNV パッケージを利用して、がんの可能性が高い細胞に
CNV スコア(予測スコア)を割り当てます。がん細胞と正常細胞を分類するだけでなく、正常細胞がどの様に徐々にがん細胞へと変化するかを調べることもできます。 |
|
|
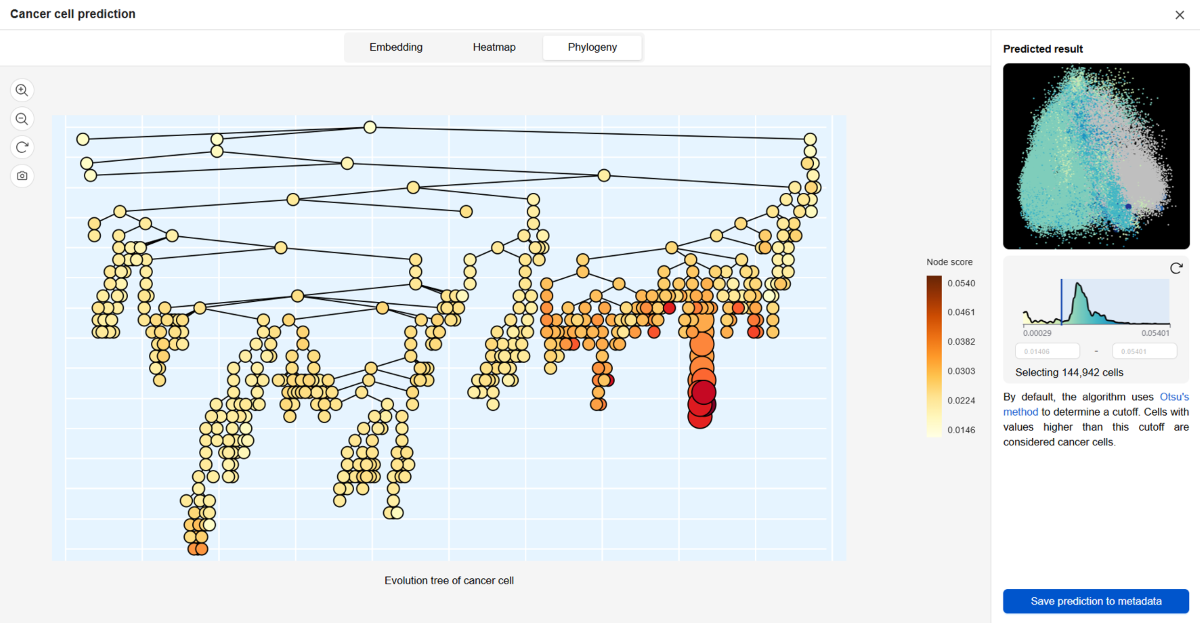
がん細胞の予測
(クリックして拡大) |
◆複数のモダリティの可視化
| |
BBrowserX Pro は、1 つの t-SNE や UMAP 上で複数のモダリティの発現情報を同時に可視化することが可能です。以下の様なデータに対応しています。
・scRNA-seq: 遺伝子発現情報
・Perturb-seq: gRNA 発現情報
・CITE-seq: 細胞表面タンパク質発現情報 |
|
◆Differential expression
| |
特定の 2 つのクラスターとそれらの関連付けられているプロセスの間で発現に差異がある遺伝子を特定できます。ボルケーノプロットを備えたインタラクティブなダッシュボードで視覚的な比較が可能です。
さらに、得られた結果からエンリッチメント解析を実行することで、それらの遺伝子の背後にある生物学的メカニズムを明らかにすることができます。 |
|
|

Differential expression
(クリックして拡大) |
◆Marker genes
| |
ある細胞集団を他の細胞集団と区別するのに役立つ遺伝子を見つけることができる機能です。 |
|
◆Pseudotime(擬似時系列解析)
| |
Pseudotime analysis は、遺伝子発現プロファイルの動的な変化をモデル化することで、細胞の発生や分化の軌跡を再構築する計算的手法です。 |
|
|
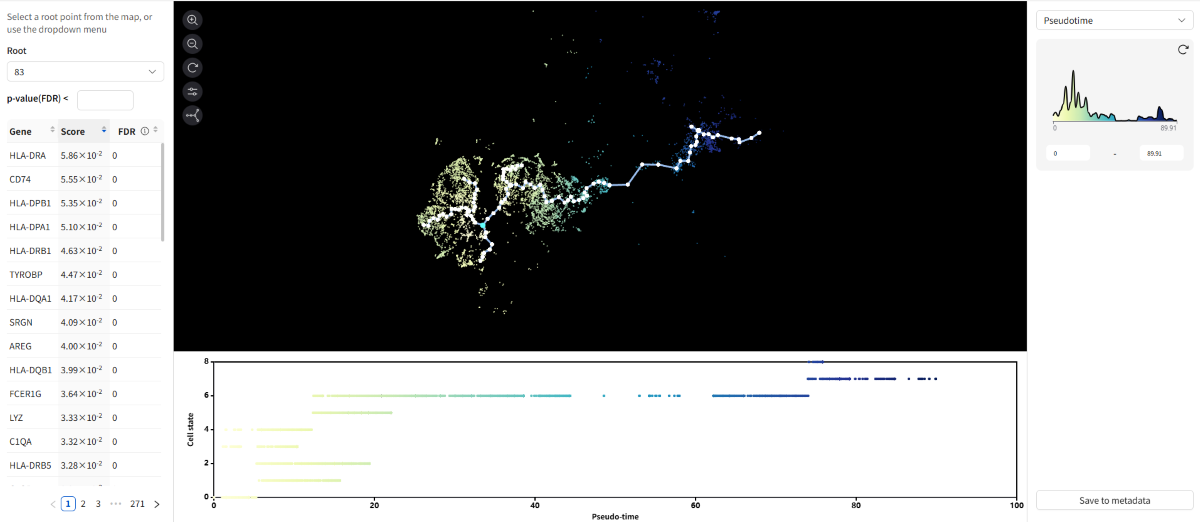
Pseudotime
(クリックして拡大) |
◆細胞間コミュニケーションの検出(Cell-cell communication)
| |
細胞シグナル伝達は、細胞間レベルと細胞内レベルの両方で極めて複雑なネットワークがあり、細胞周期の活性化、プログラム細胞死、細胞移動、分化といった様々な細胞プロセスを理解する上で極めて重要な役割を果たしています。
シングルセルデータには、遺伝子発現情報が含まれており、これを利用することで細胞間コミュニケーションを推定することができます。 |
◆サブクラスタリング
| |
特定の細胞群を抽出し、それらの新たなデータセットとして再解析する高度な手法です。元のデータ全体では見落とされがちな、細胞群に特有の構成要素や重要な特徴をより詳細に捉えることが可能です。 |
|
|
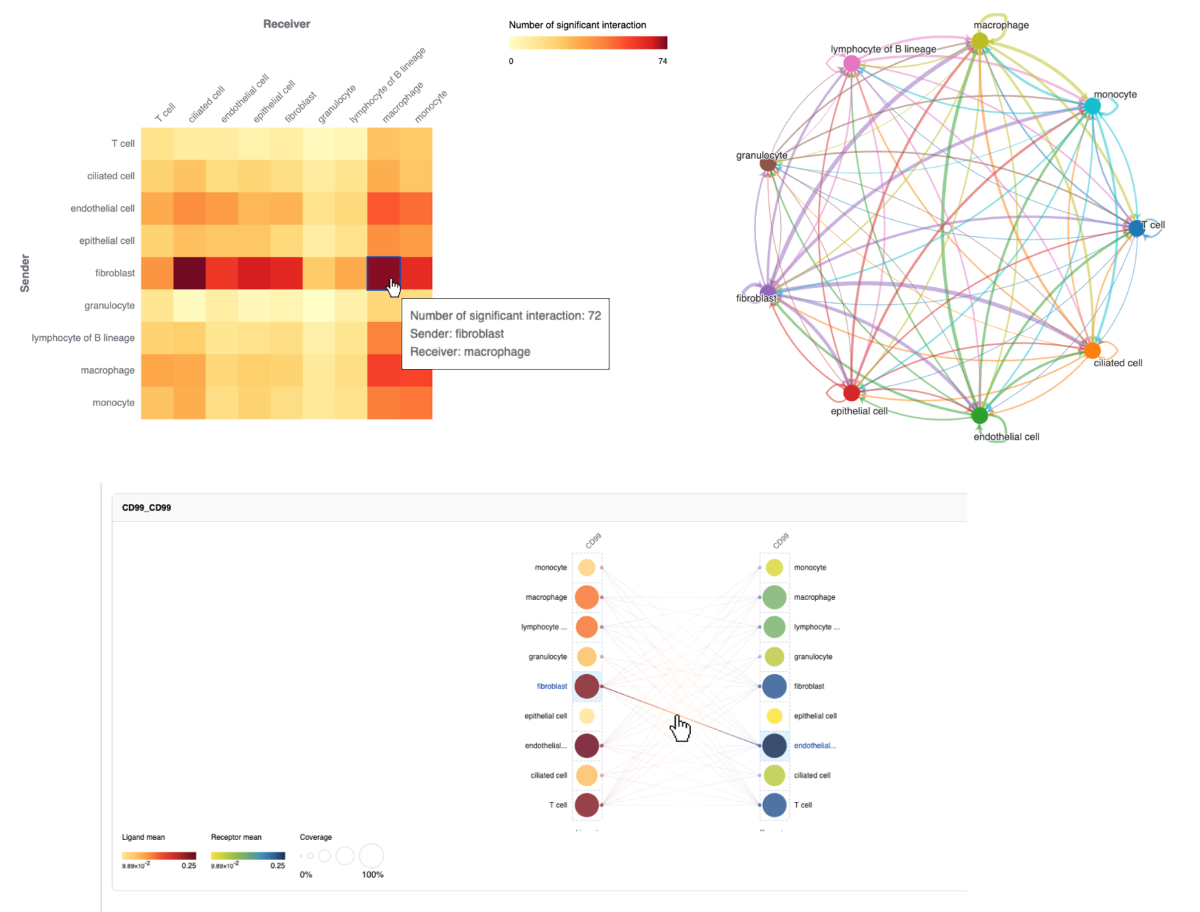
Cell-cell communication
(クリックして拡大) |
◆多彩な視覚化
| |
バイオリンプロット、ボックスプロット、バブルヒートマップ、ヒートマップ、UpSet プロットなどの様々なグラフを使用して、グループ間の遺伝子発現パターンを調べることができます。Composition
Chart では、複数の条件における細胞の組成と遺伝子発現を視覚化できます。例)各サンプルにおける細胞種の割合 |
|
|
 デモ版ダウンロード デモ版ダウンロード

|
弊社( )までお問合せください。 )までお問合せください。 |
 システム要件/サポートされる生物種 システム要件/サポートされる生物種

|
ウェブブラウザ上で動作します。
計算・データ保存は、メーカーのクラウド上で行われます。
インターネット接続を必要とします(Google Chrome 推奨)
接続について、デモ版にてテストすることを推奨しております。
生物種は、ヒトおよびマウスがサポートされています。 |
 技術資料・セミナー 技術資料・セミナー

|
フィルジェンWEBセミナー
| シングルセル RNA-seq ・ 空間オミックスデータを用いた細胞間コミュニケーション解析(2026年2月19日) |
 |
| 免疫細胞の役割を紐解く scRNA-seq データ解析入門(2025年9月11日) |
 |
| シングルセル RNA-Seq 解析の細胞種同定(アノテーション)(2024年9月18日) |
 |
| はじめてでもできるシングルセルRNA-Seqデータ解析(2023年11月21日) |
 |
|
 価格 価格

|
アカデミックライセンス
| 商品名 |
ライセンスタイプ |
税別価格 |
カタログ# |
| BBrowserX Pro |
アカデミック、1年間 1ユーザー 指定ライセンス |
お問合せ |
F-BTX |
コマーシャル
| 商品名 |
ライセンスタイプ |
税別価格 |
カタログ# |
| BioTuring ecosystem |
コマーシャル、年間ライセンス |
お問合せ |
お問合せ |
| * |
本ソフトウェアは、年間ライセンスです。 |
| * |
解析にはインターネット接続が必要です。 |
| * |
テクニカルサポートは、電話、あるいは、電子メールでの対応のみです。 |
| * |
30GBストレージ制限があります(アカデミックライセンス)。 |
| * |
アカデミックライセンスには、グループ共有機能は付属しません。 |
| * |
BioTuring社製品を複数モジュール組み合わせてご購入頂く場合は、ディスカウント致します。 |
| * |
共通機器利用向けのライセンスもございます。詳細はお問合せください。 |
| * |
コマーシャルライセンスの仕様については、お問合せください。 |
|
|