製品・サービス情報
|
|
DiaCarta社製品
核酸抽出が不要な HPV E6/E7 mRNA 検出キット |
|
 |
 核酸抽出が不要な HPV E6/E7 mRNA 検出キット 核酸抽出が不要な HPV E6/E7 mRNA 検出キット

|
本製品は、独自の技術である isobDNATM テクノロジーを用いた HPV E6/E7 mRNA を定性検出するキットです。核酸精製、逆転写反応、ターゲット RNA 増幅が不要なため、シンプルかつハイスループットな解析を実現しています。HPV
感染のスクリーニングと予後診断の研究に有用であり、子宮頸がんと頭頸部がん用の検出キットをそれぞれラインアップしています。
【HPV E6/E7 mRNA レベルの測定について】
|
HPV E6/E7 発現レベルは、子宮頸部等のがん進行を確認する重要なバイオマーカーとしての役割を果たすことが実証されています。E6/E7 DNA
測定は、HPV E6/E7 mRNA と比較すると、一過性感染と持続性感染を区別できないため、偽陽性の結果を取得することに繋がります。HPV
E6/E7 mRNA レベルの検出は、子宮頸がんの予後をより正確に評価することが可能です。 |
【ワークフロー】
|
独自の isobDNATM テクノロジーを用いたサンドイッチ核酸ハイブリダイゼーション法を用いることにより、サンプルから直接 14 の高リスク HPV サブタイプすべての
E6/E7 mRNA を同時に検出することが可能です。本テクノロジーは、標的特異的ヌクレオチドプローブによって、E6/E7 mRNA をキャプチャし、その後シグナル増幅用のラベルプローブをハイブリダイゼーションします(下記ステップ 2-5)。その後ルミノメーター等を用いて蛍光値を検出します。
⇒isobDNATM テクノロジーの詳細
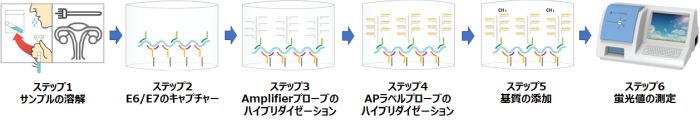
はじめに 14 の HPV 高リスクサブタイプの E6/E7 mRNA 検出を行います。検出されたサンプルについては、HPV16 および 18
のジェノタイピングを実施し、サンプルが HPV16 または/および 18 であるか、またはそのいずれでもないかを特定することが可能です。必要に応じて、14
の高リスク HPV サブタイプ検出のみ実施するか、HPV 16 および 18 のジェノタイピングのみを実施するかを選択することが可能です。 |
【QuantiVirusTM HPV E6/E7 mRNA Test の利点】
|
| ・ |
核酸精製、逆転写反応、ターゲット RNA 増幅が不要 |
|
・ |
持続感染と一過性感染を区別可能 |
| ・ |
高い感度(>90%)と優れた特異性(>90%) |
|
・ |
コルポスコピーの一致率の向上 |
|
【製品仕様】
|
本製品は、サンプルタイプによって、2種類の仕様から製品をお選びいただけます。
| 製品名 |
QuantiVirusDNA® HPV E6/E7 mRNA Test
for Cervical Cancer |
QuantiVirus® HPV E6/E7 mRNA Test
for Head and Neck Cancer |
| 品番 |
DC-01-0001R |
DC-01-0002R |
| 容量 |
96反応分 |
96反応分 |
| サンプルタイプ |
パップスメアまたは液体ベースの細胞診法によって収集されたサンプル等 |
口腔スワブサンプル、唾液サンプル等 |
| 測定機器 |
ルミノメーター
要件:420nm の波長読み取り
読み取り時間が0.2秒/ウェル
検証済み機器:
・DiaCarta Luminometer
・SpectraMax L Microplate Reader (Molecular Device Inc.)
※420nm 波長測定可能なルミノメーターやマイクロプレートリーダーをご使用いただけます。 |
| 税別価格 |
お問合せ |
| マニュアル |
[PDF] |
[PDF] |
| データシート |
[PDF] |
|
|
 注意事項 注意事項

|
・ |
本キットは、研究用として販売しております。ヒト、動物への医療・臨床診断用には使用しない様にご注意ください。また、食品・化粧品・家庭用品などとして、使用しないでください。 |
| ・ |
無断で承認を得ず、製品の再販・譲渡、再販・譲渡のための改変、商用製品の製造に使用することは禁止されています。 |
|