製品・サービス情報
|
|
Arraystar社 RNAメチル化アレイ
m6A Single Nucleotide Array 受託解析サービス |
|
 |
本サービスは、メチル化感受性 MazF RNase に基づくマイクロアレイを使用することで、単一ヌクレオチドの解像度で正確なm6A部位を特定し、その修飾の割合を定量化することができます。この手法は、他の技術で類似した解析を行うことはできないため、本サービスを利用することで、m6Aの分子メカニズムや生化学的機能、疾患の関連性に関する研究の強化につながります。
 概要 概要

|
本解析に使用するマイクロアレイは、MeRIPなどのm6A抗体による免疫沈降ベースの解析アプローチとは異なるプロファイリング方法を採用しています。100~200ntほどの解像度が限界の免疫沈降ベースのシークエンスと比較して、MazF
RNaseに基づくこのアレイは、単一ヌクレオチドの解像度で、m6ACAの正確な検出を実現しています。m6A修飾の割合あるいはパーセンテージは、各詮索部位で定量化され、動的なm6A状態を決定する際の新たなアプローチに対応しています。
また、ある程度のサンプル量が必要となるシークエンス解析と比較し、本アレイはわずか数μgのtotal RNAで解析が可能なため、少量のRNAサンプルでプロファイリングを行うことができるため、限りあるサンプルや希少な病理標本、低収量な特定の組織部位や細胞、あるいは小動物モデルで解析が可能となります。
【信頼性の高いコレクションと体系的なアノテーション】
|
Arraystar社では、Single-ACA、Poly-ACAまたは、Cluster-ACAにおける定量化可能なm6A部位を収集し、アノテーションを行うための独自のパイプラインを確立しています。
多くのm6A修飾は、コアACA配列を持つm6Aモチーフで発生し、それらはm6ACA部位と呼ばれています。ほとんどのm6ACA部位は、単一ヌクレオチドの解像度でm6Aメチル化のプロファイリングが可能なSingle-ACAで構成されています。また、複数のACA、あるいはクラスター化されたACAを持つm6ACA領域も合わせてプロファイリングされます。
◆定量化可能なSingle-m6ACA部位のコレクション - 40nt未満に別のACAが存在しないACA部位
|
本解析で定量化されるSingle-m6ACA部位は、以下の2ステップを経てコレクションされています。
(ステップ1)
本解析では、隣接したACA部位が少なくとも40nt離れているACA部位を、定量化可能なSingle-ACA部位として定義しています。m6ACA部位は、中央に位置するACA配列を基準にデザインされたプローブを使用しハイブリダイゼーションします。中央に位置する
ACA部位がメチル化されていなかった場合、MazF RNaseがACAを切断するため、プローブの結合が妨げられる、あるいは大幅に低減され、これによってメチル化レベルを定量化することを可能にしています。
近くに別のACA部位があると解析対象部位の正確な検出や定量化に影響が出る可能性がありますが、解析対象部位から40nt以上離れている場合、隣接するACA部位のプローブのシグナルの影響がないため、この性質を利用してSingle-ACA部位のm6A修飾を単一ヌクレオチドの解像度で定量的にプロファイリングを行うことができます。Arraystar社では、最新のRefseqデータベースのトランスクリプトシークエンスに基づいてすべての定量化可能なSingle-ACA部位をコレクションしています。
|
|
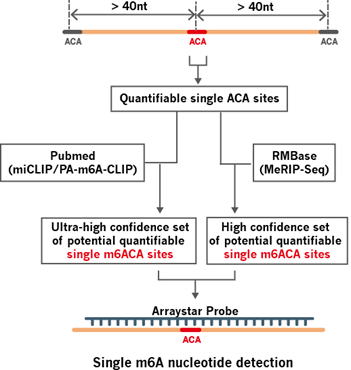
図1. Single-m6ACA部位の収集パイプライン |
(ステップ2)
RNAのすべてのACA配列がm6Aによって修飾されるわけではありません。ステップ1で収集されたすべての定量化可能なSingle-ACA部位はmiCLIPデータセットに集約された実際のm6A部位、またはm6A-seqピークに近いm6Aコンセンサスモチーフにマッピングされ、これらはそれぞれ非常に高い信頼性のSingle-ACAセットを定義しています。これらのSingle-m6ACA部位が、最終的にArraystar社のm6A
Single Nucleotide Arrayに集約されています。
|
|
◆定量化可能なPoly-m6ACA領域のコレクション - 20nt未満の領域に近接して配置された複数のACA部位
|
本解析で定量化されるPoly-m6ACA部位は、以下の2ステップを経てコレクションされています。
(ステップ1)
本解析では、RNA配列の20nt未満に複数のACA部位が含まれている領域を、定量化可能なPoly-ACA領域として定義しています。それぞれ単一のACAのm6A修飾を解析する代わりに、Poly-ACAは領域をカバーするプローブによって共同で検出と定量化が行われます。
つまり、プローブでカバーされているACAのいずれかがメチル化されておらず、MazF RNaseによって切断された場合、プローブのシグナルが影響を受けます。そのため、プローブのシグナルが得られた場合、領域内のすべてのACAがメチル化されていることを示します。
マイクロアレイによって検出されたPoly-ACA部位は、単一ヌクレオチドではありませんが、20nt未満の非常に高い解像度として、これらすべての定量可能なPoly-ACA領域をコレクションしています。
|
|
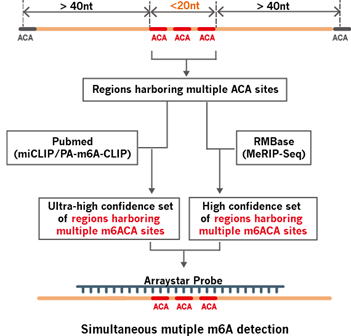
図2. Poly-m6ACA部位の収集パイプライン |
(ステップ2)
RNAのすべてのACA配列がm6Aによって修飾されるわけではありません。ステップ1で収集されたすべての定量化可能なPoly-ACA領域はmiCLIPデータセットに集約された実際のm6A部位、またはm6A-seqピークに近いm6Aコンセンサスモチーフにマッピングされ、これらはそれぞれ非常に高い信頼性のPoly-ACAセットを定義します。これらのPoly-m6ACA領域が、最終的にArraystar社のm6A
Single Nucleotide Arrayに集約されています。 |
|
◆定量化可能なCluster-m6ACA領域のコレクション
|
複数のSingle-ACA部位、あるいはPoly-ACA領域が500nt未満に含まれ、それらの距離が100nt未満の場合、それらを1つのクラスター化されたACA領域として定義しています。これは、構成要素となるSingle-ACA部位およびPoly-ACA領域の個々のプローブのシグナルを統合することにより解析を行います。すべてのプローブからシグナルが得られた場合、クラスター内のすべてのm6ACA部位がメチル化されていることを示しています。
|
|
【単一およびポリメチル化m6A部位の機能的な重要性】
|
mRNAには平均して約3つのm6A修飾があります。多くのm6A修飾mRNAには、Single-m6A部位しか含まれていませんが、一部のmRNAには20以上の部位が含まれている可能性があることが知らされています。Single-m6A部位は、上流と下流のどちらの場合でも近くに他のm6A部位はありませんが、Poly-m6A領域の場合は、隣接するm6A部位が短い領域内に集約される傾向にあることが知らされています。これらの単一およびポリメチル化されたm6A修飾は、分子、細胞、および生物の表現型にとって機能的な重要性を持っています。
◆細胞および生物の表現型に対するSingle-m6A部位の機能的重要性
|
単一のm6A修飾は、mRNAの翻訳開始、または翻訳伸長の動態における分子機能に関係している場合が多くあります。これらはまた、non-coding
RNAの減衰や活性を調節しています。
これらの重要性に関しての詳細情報は、Arraystar社のwebサイトからご確認頂けます。 ⇒こちら |
|
|
|
 サービスワークフロー サービスワークフロー

|
MazF RNaseは、メチル化されたm6ACA配列ではなく、メチル化されていないACA配列の5'末端側の一本鎖RNAを選択的に切断する酵素特異性を持っています(図1)。
このMazF消化に基づいて処理されたm6ACAにより切断されていないRNA断片と、MazF RNase未処理のinput RNAを2色で蛍光標識し、Arraystar社のm6A
Single Nucleotide Arrayとハイブリダイズ、単一ヌクレオチドの分解能でm6Aレベルの定量値を取得します。この方法は、m6A抗体による免疫沈降を使用した半定量的な解析と比較して高精度に定量化が可能です。 |
|
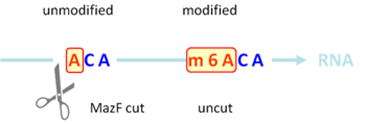 |
|
|
【Arraystar社のサービスの作業手順】
|
・DNaseによるサンプルのDNA処理
・MazF消化による未修飾ACA配列の切断(input RNA サンプル及びMazF処理サンプルの作製)
・ハイブリダイゼーションシグナル正規化のためのリファレンスRNAのスパイク
・input RNAサンプルをCy3標識、MazF処理サンプルをCy5標識
・Cy3およびCy5 cRNA混合物のアレイハイブリダイゼーション
・アレイスキャンニングおよびデータ解析 |
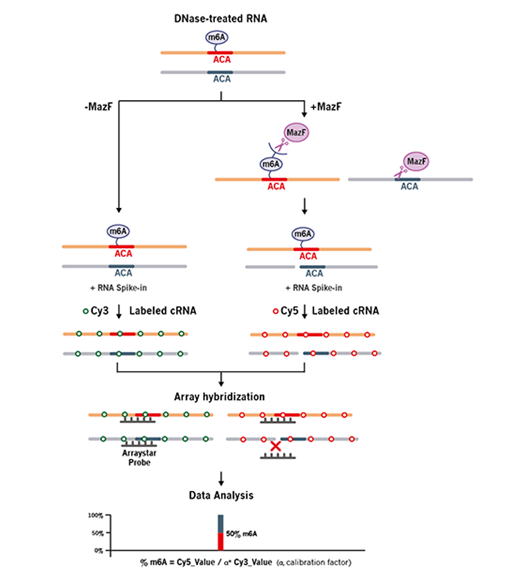
【データ解析について】
|
Arraystar社で実施される豊富で詳細なバイオインフォマティクス解析とアノテーションには、m6A修飾化学量論、m6A存在量レベル、単一ヌクレオチド解像度でのm6A位置、転写モデル領域などが含まれています。これらは、m6Aの動力学、生物学的機能、分子メカニズム、および疾患の関連性を理解するために不可欠ですが、他の解析手法では利用することができません。
・m6Aメチル化の差異
比較条件/グループ間の異なるm6A部位メチル化化学量論と異なるm6Aサイト存在量の両方が提供されます。 |
|
|
 マイクロアレイ情報 マイクロアレイ情報

|
本サービスでは、解析に使用するマイクロアレイを、ヒト、マウス、ラットの3生物種でご用意しています。
| Human m6A Single Nucleotide Array |
| プローブ総数 |
14,321 |
| プローブ長 |
60nt |
| Single-ACA部位 |
11,237 |
| Poly-ACA領域 |
3,084 |
| Cluster-ACA領域 |
693 |
| m6ACA部位のソース |
miCLIP dataset (Linder et al., 2015; Ke et al., 2015; Xu et al., 2017; Chen et al., 2015)
RMbase database (Schwartz et al., 2013) |
| アレイフォーマット |
8x15K |
| Mouse m6A Single Nucleotide Array |
| プローブ総数 |
14,319 |
| プローブ長 |
60nt |
| Single-ACA部位 |
11,120 |
| Poly-ACA領域 |
3,199 |
| Cluster-ACA領域 |
279 |
| m6ACA部位のソース |
miCLIP dataset (Linder et al., 2015; Ke et al., 2015; Xu et al., 2017; Chen et al., 2015)
RMbase database (Schwartz et al., 2013) |
| アレイフォーマット |
8x15K |
| Rat m6A Single Nucleotide Array |
| プローブ総数 |
14,581 |
| プローブ長 |
60nt |
| Single-ACA部位 |
11,499 |
| Poly-ACA領域 |
3,082 |
| Cluster-ACA領域 |
2,614 |
| m6ACA部位のソース |
Mouse M6ACA site homologs (Linder et al., 2015; Ke et al., 2015; Xu et al., 2017; Chen et al., 2015)
RMbase database (Schwartz et al., 2013) |
| アレイフォーマット |
8x15K |
【データ解析】
|
本サービスは、マイクロアレイからバイオインフォマティクス解析までがパッケージとなっております。バイオインフォマティクス解析に関しては、ご依頼頂いた全てのお客様に実施させていただくスタンダード解析と、オプションで追加できるアドバンス解析(別途追加費用が発生いたします)をご用意しております。
◆スタンダード解析
|
本解析では、m6A修飾化学量論、m6A存在レベル、単一ヌクレオチド分解能でのm6A位置、転写モデル領域などの豊富なバイオインフォマティクス解析およびアノテーションをご提供いたします。
1. 差異的なm6Aメチル化情報
|
比較条件/グループ間の異なるm6A部位のメチル化化学量論およびm6A部位の存在量に関する情報をご提供いたします。
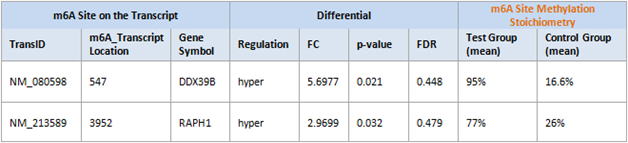
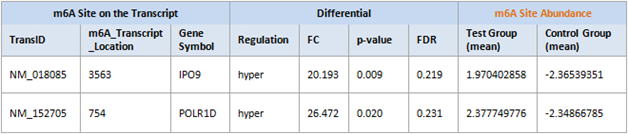
|
2. m6A部位の体系的なアノテーション情報
|
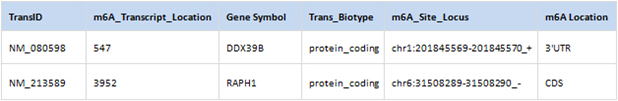
| ProbeID: |
Probe ID. |
| TransID: |
Database accession of the transcript being m6A modified. |
| Trans_biotype: |
Transcript biotype:"ncRNA" for noncoding RNA or "protein_coding" for mRNA. |
| Gene_Symbol: |
Official gene symbol for the transcript host gene. |
| m6A_transcript_location: |
m6A location in the transcript. |
| m6A_site_Locus: |
Genomic coordinates of the m6A site. |
| m6A_location: |
Transcript model region where the m6A site is located: 5'UTR, CDS or 3'UTR. |
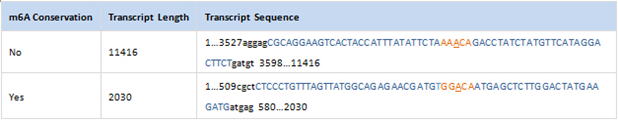
| m6A_conservation: |
If the m6A site has conserved orthologous m6A site between human and mouse species. |
| Transcript_Length: |
Full length of the transcript. |
| Transcript_Sequence: |
Sequence of the transcript, where, Orange = m6A motif; Orange and underlined
= m6A position; Uppercase = probe sequence; |
|
|
◆アドバンス解析(別途追加料金が発生いたします。)
|
m6ACA部位は転写部位に単一(Single-)、複数(Poly-)、およびクラスター(Cluster-)化された部位パターンを構成することができます。Single-m6ACA部位に加えて、複数またはクラスター化されたm6A部位が高解像度で連帯的にプロファイルされ、体系的にアノテーション付けられます。
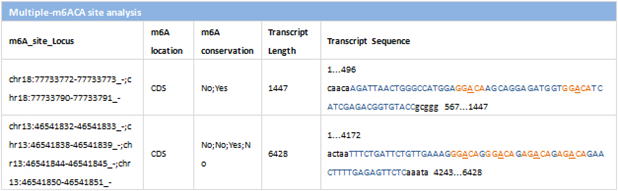
| m6A_site_Locus: |
List of genomic coorinates of the constituent sites in the multiple-m6ACA
site, separated by a semicolon. |
| m6A_location: |
Transcript model region where the multiple-m6A site is located: 5'UTR,
CDS or 3'UTR. |
| m6A_conservation: |
If the multiple-m6ACA site has conserved orthologous site between human
and mouse species. |
| Transcript_Length: |
Full length of the transcript. |
| Transcript_Sequence: |
Sequence of the transcript, where, Orange = m6A motif; Underlined = m6A
position; Uppercase = probe sequence; |
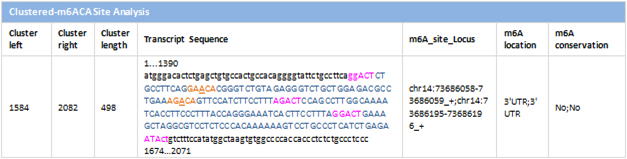
|
| Cluster_left: |
Left boundary of the m6A cluster in the transcript. |
| Cluster_right: |
Right boundary of the m6A cluster in the transcript. |
| Cluster_length: |
Length of the m6A cluster. |
| Transcript_Sequence: |
Sequence of the transcript, where,
Orange = MazF cleavable m6A motif, for probe detection;
Pink = MazF uncleavable m6A motif, for defining m6A cluster only;
Underlined = m6A position;
Uppercase = Cluster sequence, which starts and ends with m6A positions
in the first and the last m6A motifs. |
| m6A_site_Locus: |
List of genomic coordinates of the m6A sites, separated by a semicolon. |
| m6A_location: |
List of transcript model region where each constituent m6ACA site resides
(5'UTR, CDS or 3'UTR), separeted by a semicolon. |
| m6A_conservation: |
If the m6A site has conserved orthologous m6A site between human and mouse
species. |
|
|
 サンプル条件 サンプル条件

|
| 生物種 |
サンプルタイプ |
必要量・濃度 |
RIN値 |
純度 |
| ヒト、マウス、ラット |
Total RNA |
≧5μg (≧0.1μg/μL) |
≧7.0 |
O.D.260/280: ≧1.8
O.D.260/230: ≧1.8 |
| 1. |
電気泳動写真の添付を推奨します。
※電気泳動は、次の条件を推奨します。1.0%アガロースゲル; 1.0%TAEバッファー; 100V, 40min. |
| 2. |
DNase, RNaseフリーの1.5mLまたは2mLのチューブを使用してください。(スクリューキャップ式を推奨) |
| 3. |
total RNA は、RNaseフリー水(ddH2Oなど)に溶解し、-80℃で保存してください。 |
| 4. |
DNase処理を行うことを強く推奨します。 |
| 5. |
UVスペクトルベースの濃度測定法では、不正確になる場合がありますので、蛍光ベースの測定を強く推奨します。
※UVスペクトルベースで測定した場合、上記よりも多いRNAを準備してください。 |
| 6. |
スピンカラムでRNA抽出・精製する際、低分子のRNAなどが除去されることがあるため、必ずキットの仕様を確認してください。 |
| 7. |
上記のサンプル条件は、ご準備頂く際の目安であり、最終的な品質は業務提携先でのQCチェックの結果に従います。 |
|
 価格 価格

|
| サービス名 |
生物種 |
税別価格 |
カタログ# |
m6A Single Nucleotide Array
受託解析サービス |
Human |
¥246,000 |
F-AS-SNAH-[サンプル数] |
| Mouse |
F-AS-SNAM-[サンプル数] |
| Rat |
F-AS-SNAR-[サンプル数] |
|
 ご注意事項(必ずご確認ください) ご注意事項(必ずご確認ください)

|
| 1. |
解析サービスに関して
本サービスの解析は、弊社の海外提携先(Arraystar社)にて実施されます。 |
| 2. |
解析に利用する製品に関して
本サービスで使用するマイクロアレイは、事前の予告なくバージョンアップされる場合があります。 |
| 3. |
サンプルの送付および取り扱いに関して
| 3.1 |
|
サンプルのご発送は、必ず、サンプル送付方法およびご注意点(PDF)の内容に従ってください。送付の際は、チューブをパラフィルムで覆い、キャップの緩みや中身の漏れがおきない様にしてください。サンプルの入ったチューブには、油性マジックでサンプル名を明記し、ビニールバッグ等に入れてください。RNAサンプルは、十分量の砕いたドライアイスとともに梱包し、冷凍便でお送りください。 |
| 3.2 |
|
サンプルは、なるべく休日をはさまない様に、弊社営業時間内(土日祝祭日を除く、平日9時~18時)に到着する様にご発送ください。なお、弊社までの送料は荷送り人様負担とさせて頂いておりますので、あらかじめご注意ください。 |
| 3.3 |
|
サンプル送付の際は、QCシートおよび免責事項同意書を同封してください。これらが同封されていない、または記入漏れがある場合、解析サービスには着手できませんので、ご注意ください。 |
| 3.4 |
|
ご送付頂いたRNAの品質チェックの結果、解析に必要な量が不足または低品質であった場合、サンプルの再送をお願いする場合があります。なお、再度サンプルをお送り頂く場合、提携先への再送料金をご負担頂きます。 |
| 3.5 |
|
弊社に起因しない(国内および国際輸送時など)サンプル喪失等に対する補償・保険制度はありませんので、貴重なサンプルをご提供頂く際はご注意ください。 |
| 3.6 |
|
お預かりできるサンプルは、BSL2(バイオセーフティーレベル2)までに限られます。感染性が著しく高いサンプル(HIV、HCV、HBVなどに感染している検体など)は、お預かりできません。ヒト臨床サンプルの場合、インフォームドコンセントを得てからご提供ください。 |
| 3.7 |
|
ご提供頂いたサンプルの返却は致しておりません。(サンプルは解析終了時に破棄されます。) |
| 3.8 |
|
本確認事項を満たさない事で、別途費用が発生した場合、お客様に費用のご負担をお願いすることがあります。 |
| 3.9 |
|
サンプル・業務等から生じた知的財産権・工業所有権・安全性・インフォームドコンセント等の問題について、弊社は一切の責任を負わないものとします。 |
|
| 4. |
キャンセルに関して
本サービスは、海外での解析という性質上、サンプル受領後のキャンセルはお引き受けできません。やむを得ない理由でキャンセルされる場合、それまでの工程に応じた料金をご請求いたします。 |
| 5. |
納品物に関して
納品データのバックアップは、必ずお客様にてお取りください。弊社でのデータ保管期間は、発送日から90日間です。弊社での保管期間を経過したデータは破棄されます。 |
|
*本サイトの情報は、Arraystar社のウェブサイトを一部引用しております。
|