 概要 概要

|
|
CLC MLST Moduleは、バクテリアや微生物の亜種決定及び解析を行うためのMLST(Multi Locus Sequence Typing)専用の解析モジュールです。グラフィカルなユーザーインターフェースで容易に解析を行うことができます。MLSTは、バクテリアや酵母菌から分離されたものを分類するためのポータブルで精密な最先端技術です。その多くがハウスキーピング遺伝子からのDNA配列データと既知の対立遺伝子座に関するデータベースと比較により行われ、すべての遺伝子の統合情報から最終的な配列タイプを決定します。
本ソフトウェアは、以下のソフトウェアにPlug-inして使用するものです。従いまして、以下のソフトウェアも必要となります。
・CLC Genomics Workbench
・CLC Main Workbench |
|
 簡単!ユーザーフレンドリー 簡単!ユーザーフレンドリー
 |
 |
|
CLC MLST Moduleでは、解析する配列データを選択し、MLSTスキームを実行するだけで数秒後には配列タイプが決定されます。データの修正・編集、結果の確認も簡単に実行できます。 |
|
 時間短縮 時間短縮
 |
|
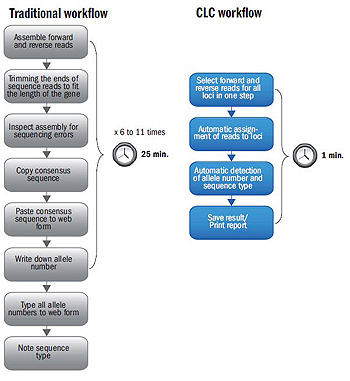
通常のMLST作業フローでは、アセンブル、アセンブルの確認、配列末端のトリミング、ウェブフォームへのコピー&ペースト、対立遺伝子座番号の書き込みなどが含まれています。すべての遺伝子座について、この作業を繰り返す必要があり、平均で25分かかります。CLC
MLST Moduleを使えば、このワークフローは1分未満で完了できます。 |
|
 反復ワークフロー 反復ワークフロー
 |
|
ある遺伝子座のシークエンシング実験で失敗したとしても、他の成功した実験より、配列タイプの予備決定をすることができます。また、再実験後に失敗した遺伝子座のデータを加え、再解析することができます。この様に配列タイプの決定におけるあらゆる段階でデータ修正と補整をすることができます。 |
|
 結果の保存 結果の保存
 |
|
CLC MLST Moduleでは、すべての作業を簡単に保存し、ワンクリックするだけで、元に戻すことができたり、結果を補正することができます。同時にヒストリービューを使えば、ユーザーのすべての分析を記録し、文書化したり、分析を繰り返したり、改良することが簡単に行えます。 |
|
 ユーザー独自のスキーム拡張・開発 ユーザー独自のスキーム拡張・開発
 |
 |
|
CLC MLST Moduleは、PubMLST、MLST.netやMax-Planck-Institut fur Infektionsbiologieデータベースから、MLSTスキームを簡単にダウンロードすることが可能です。ダウンロード後に、ユーザーは、新たなスキームや独自の対立遺伝子座タイプを加えることによって、自由にスキームをカスタマイズできます。 |
|
 総合的ソリューション 総合的ソリューション
 |
 |
|
CLC MLST Moduleは、CLC Genomics Workbenchや、CLC Main Workbenchをスムーズに統合するプラグインです。これにより、系統発生、プライマーデザイン、BLAST、SNP注釈、及びバーチャルクローニングなどのすべてのシークエンシング解析作業を完全統合することが出来ます。 |
|
 価格 価格
 |
|
| 商品名 |
ライセンスタイプ |
税別価格 |
カタログ# |
| CLC MLST Module |
固定年間ライセンス/アカデミック |
お問合せ |
お問合せ |
| 固定年間ライセンス/コマーシャル |
お問合せ |
お問合せ |
| ネットワーク年間ライセンス/アカデミック |
お問合せ |
お問合せ |
| ネットワーク年間ライセンス/コマーシャル |
お問合せ |
お問合せ |
| * |
本商品は、CLC Genomics Workbench/CLC Main Workbenchの機能拡張モジュールです。本商品単独では使用できませんので、ご注意ください。 |
| * |
テクニカルサポートは、電話あるいは電子メールで対応いたします。 |
| * |
固定ライセンスは、搭載CPUの論理コア数から64コアを超えるコンピュータへのインストールと、リモート操作による使用はできません。 |
| * |
データベースからのデータのダウンロードを行うのに、インターネット接続が必要になります。 |
|
|