製品・サービス情報
(注目製品)
|
|
QIAGEN社/CLC bio製品のソフトウェア単体販売からインテグレーションまで、すべて取り扱える正規販売代理店です。
NGS Bioinformatics Solution
次世代シークエンス解析用ソフトウェア
CLC Genomics Workbench |
|
  |
 概要 概要

|
CLC Genomics Workbenchは、次世代シークエンサーから出力される膨大なデータの解析に対応した統合配列解析ソフトウェアです。
最先端の解析アルゴリズムと高速なゲノムアセンブル・マッピングツール、豊富なグラフィカル機能や多彩な出力オプションを搭載し、ユーザーフレンドリーかつ直感的なインターフェイスで稼働します。
Pacific bioscience社やillumina社、Thermo Fisher Scientific社の次世代シークエンサーデータに対応し、アセンブルやマッピング、各種アプリケーションに対応した専用解析モジュールを搭載しています。
さらに同社製品「CLC Main Workbench」のすべての解析モジュールも搭載しており、次世代シークエンサーのみならず、サンガーシークエンサーにも対応した、統合配列解析ソフトウェアとして使用できます。 |
|
|


カタログダウンロード(PDF) |
 主要なアプリケーション 主要なアプリケーション

|
|
 |
|
リシークエンシング解析 
次世代シークエンサーより取得したリード配列データを、既知のリファレンスゲノム配列にマッピングを行い、各種変異(SNV, InDel, CNV)の検出を行います。検出した変異には、アミノ酸置換情報や遺伝子名などをアノテーションとして付加することが可能です。 |
|
 |
|
トランスクリプトミクス解析 
RNA-SeqやsmallRNA解析によって、各遺伝子やsmallRNAごとの発現量データを網羅的に取得することができます。取得した発現量データは、専用ツールや無償プラグインを使用することで、サンプル間の発現変動解析やクラスタリングによる統計解析、ヒートマップやベン図作成によるグラフ表示を行うことが可能です。 |
|
 |
|
エピゲノミクス解析
クロマチン免疫沈降(ChIP)アッセイで取得したリード配列データが、ゲノム配列上のどの領域でピークを形成するかを解析することによって、転写因子とゲノム配列上の結合部位を同定します。ピーク領域近傍の遺伝子名をアノテーションとして取得するほか、無償プラグインの使用で、ヒストン修飾やバイサルファイトシークエンスの解析にも対応します。 |
|
 |
|
De novoシークエンシング解析 
リファレンスゲノム配列を使用せず、ゲノムシークエンスを行ったリード配列を直接つなげていき、コンティグ配列を作成します。作成したコンティグ配列は、原核生物ゲノムのORF予測や、BLASTによる相同性検索などに使用でき、配列決定から遺伝子機能の予測までをサポートします。 |
|
 |
|
その他の機能
解析ワークフロー作成、複数サンプルのバッチ解析、ゲノムデータのダウンロード、リード配列データのトリミング/クオリティーチェック、サンプル情報のメタデータ、各種プラグインのインストールなど |
 アプリケーションの詳細 アプリケーションの詳細

|
【リシークエンシング解析】
|
・リファレンスゲノムへのマッピング
NCBIやEnsemblなどのデータベースに登録されているリファレンスゲノムを使用し、次世代シークエンサーで取得したリード配列データのマッピングを行うことが可能です。パラメータの調整を行うことで、Insertion/Deletion領域にも正しくリード配列をマッピングすることができ、また、Paired
end や Mate pair などのペアリード配列データにも対応しています。さらに、リファレンスゲノムの設定時に、同時にゲノム上のアノテーションデータを登録することで、任意のアノテーション領域にターゲットを絞ってマッピングを行うことも可能です。 |
(クリックして拡大) |
| |
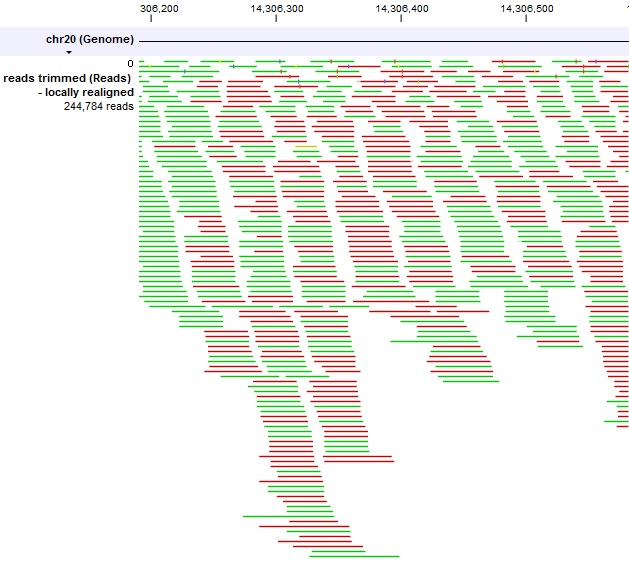
・変異検出
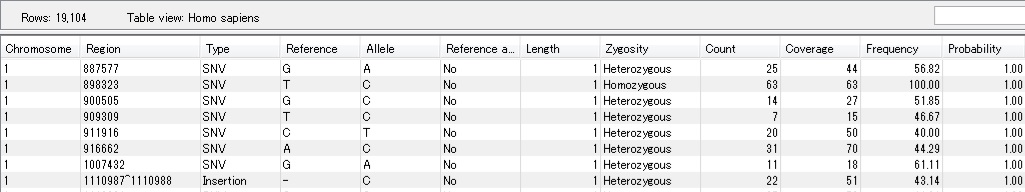
マッピングデータから変異(SNV, InDel, CNV)の検出を行うと、解析結果はテーブル形式で表示されます。テーブルには、各変異のゲノム上の位置やアリル情報、接合性情報、さらにアリルごとのリード配列のカウント情報などを含み、テーブル上でフィルターをかけることで、任意の条件を満たす変異のみをピックアップすることが可能です。また、本ソフトウェアには複数種類の変異検出用ツールが搭載されており、変異をもつリード配列のカバレッジ中の頻度や、解析サンプルの生物種の倍数性などによって、ツールを使い分けることが可能です。いずれのツールも、様々なノイズフィルターが組み込まれており、パラメータを調整することで、検出される変異の偽陽性を減らしたり、逆に検出感度を上げたりすることが可能です。 |
| |
(クリックして拡大) |
|
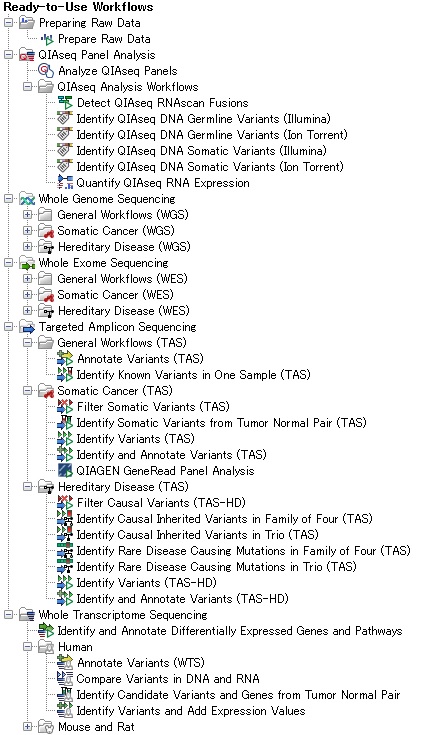
・Biomedical Genomics Analysis
無償プラグイン「Biomedical Genomics Analysis」をインストールすることで、全ゲノム解析、全エクソーム解析、ターゲットアンプリコン解析、全トランスクリプトーム解析などのヒト疾患研究用の変異解析ワークフローを使用できる様になります。自身によるワークフローの作成や、煩雑な解析手順を必要とせず、いくつかの設定を入力するだけで、直ちに解析が始まり、データを得ることが可能です。 |
(クリックして拡大) |
【トランスクリプトミクス解析】
|
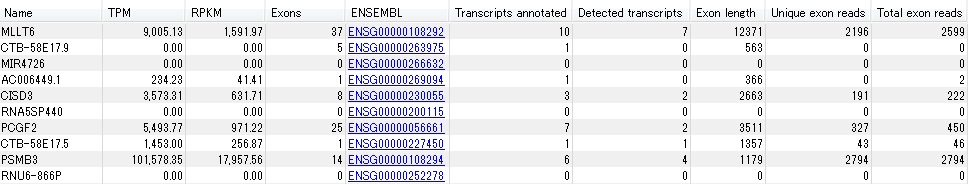
・RNA-Seq Analysis
標準搭載のRNA-Seq解析用ツールを使用すると、リード配列データのリファレンスゲノムへのマッピング、および各遺伝子の発現量の算出までを自動で行います。エクソン間をまたいだリード配列のマッピングにも対応し、遺伝子の発現量については、遺伝子ごとのリード配列カウント以外に、RPKM(Reads
Per Kilobase Million)とTPM(Transcripts Per Kilobase Million)による計算が可能です。また1つの遺伝子に複数のスプライスバリアントが存在する生物の場合は、遺伝子ごとの発現量データの他に、転写物(mRNA)ごとの発言量データも得ることができます。さらにペアリード配列データでマッピングを行う場合は、ペアとなっている各リード配列のマッピング状況から、融合遺伝子の候補を予測することが可能です。 |
|
(クリックして拡大) |
|
・Advanced RNA-Seq
RNA-Seq遺伝子発現量データのサンプル間比較解析を行うための様々なツールが使用できます。各サンプルをグループごとに分類し、グループ間で統計的に有意に発現量が変動している遺伝子の検索や、統計データによるボルケーノプロットの表示、主成分分析や二次元クラスタリングとヒートマップ、さらに複数の比較データからベン図の作成、さらにGene
Ontologyを用いた遺伝子発現解析が可能です。これらのツールでは、TMM法でデータの補正を自動的に行うので、シークエンスされたリード配列数が異なるサンプル同士のデータも、問題なく比較を行うことが可能です。 |

(クリックして拡大) |
|
・small RNA Analysis
small RNA Analysis ツールでは、small RNAのデータベースをリファレンス配列として使用し、各small RNAとmicroRNAの発現量を計算します。計算した発現量データは、専用の解析ツールを使用し、サンプル間のデータ補正と、グループ間の発現量比較を行うことが可能です。 |

(クリックして拡大) |
【エピゲノミクス解析】
|
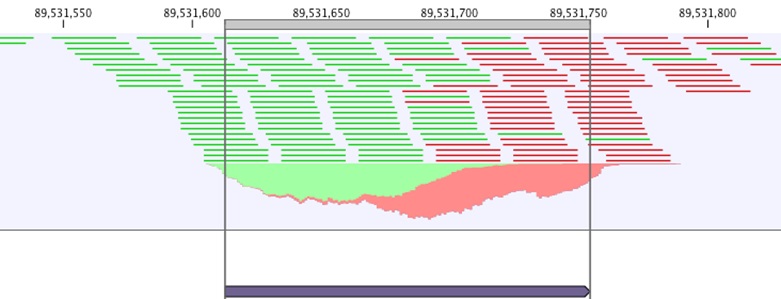
・ChIP-Seq
クロマチン免疫沈降(ChIP)アッセイと次世代シークエンスを組み合わせることで、アッセイに使用した転写因子のゲノム配列上の結合部位を同定します。シークエンスされたリード配列データを、リファレンスゲノム配列にマッピングすることでピークが形成され、専用の解析ツールを使って、ゲノム配列上のすべてのピーク領域をピックアップします。その際、ChIPサンプルとは別にInput
DNAなどのコントロールサンプルのシークエンスデータが存在する場合は、コントロールサンプルのデータを考慮して、ChIPサンプルからのピーク検出を行うことが可能です。 |
(クリックして拡大) |
|
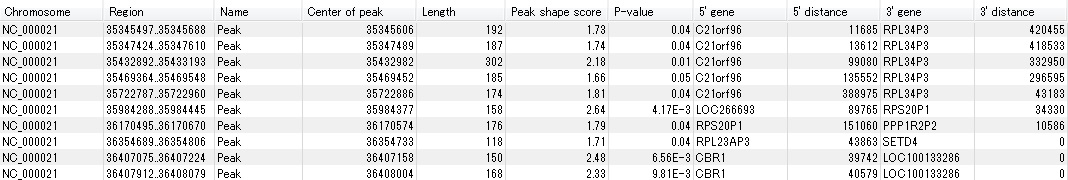
・ピーク検出
検出されたピークの情報は、テーブル形式のデータとして表示されます。テーブルには、各ピークのゲノム配列上の位置とピークの信頼性、さらにピーク近傍の遺伝子名をアノテーションとして付加することが可能です。また、無償プラグイン「Advanced
Peak Shape Tools」を使用することで、外部データに基づいたピーク検出なども行うことが可能になります。 |
|
(クリックして拡大) |
【De Novo シークエンシング解析】
|
・De Novo Assembly
De Novo Assemblyツールを使用することで、illumina社シークエンサーなどでゲノムをシークエンスして取得した、ショートリード配列データを用いたアセンブルを行い、コンティグ配列を作成することが可能です。ペアリード配列データによるScaffoldingにも対応し、コンティグ配列に自動的に付加されるアノテーション情報から、Scaffold領域などを用意に確認することができます。アセンブル実行時には、パラメータとして、Word
size(k-mer)を任意の値に設定でき、さらに解析結果のレポートも作成され、コンティグ配列データのN50値やコンティグ配列数、平均と最大コンティグ配列長、さらに全コンティグ配列の総塩基数などが記載されているため、アセンブル結果を容易に評価することもできます。 |
|

(クリックして拡大) |
|
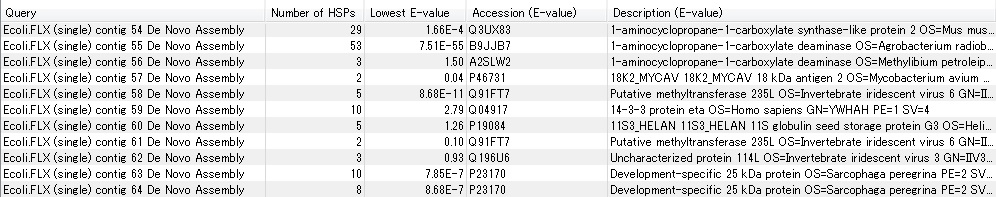
・BLAST解析
De Novo Assemblyで作成したコンティグ配列データなど、各種配列データを使用して、BLASTによるデータベースの相同性検索を行うことが可能です。解析結果はテーブル形式で表示され、各コンティグ配列と最も相同性の高かったデータを、容易に確認することが可能です。 |
|
(クリックして拡大) |
【その他の機能】
|
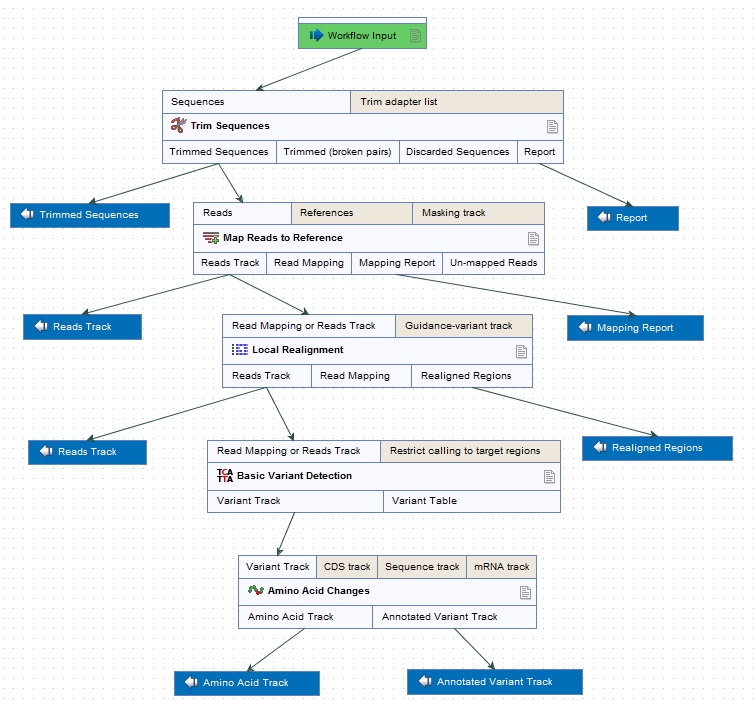
・解析ワークフロー作成
複数のツールを組み合わせて解析を行う場合、使用するツールの順番などをワークフローとして登録することが可能です。各ツールのパラメータも任意に設定でき、データ解析をルーチン的に行う場合の解析パイプラインとして使用することができます。 |
|
・複数サンプルのバッチ解析
複数のサンプルデータが存在する場合、同一の解析条件で、全サンプルを一括して解析を行うことができます。ワークフロー機能と組み合わせることで、データ解析の作業効率を大幅に向上させることが可能です。 |
|
(クリックして拡大) |
|
(クリックして拡大) |
|
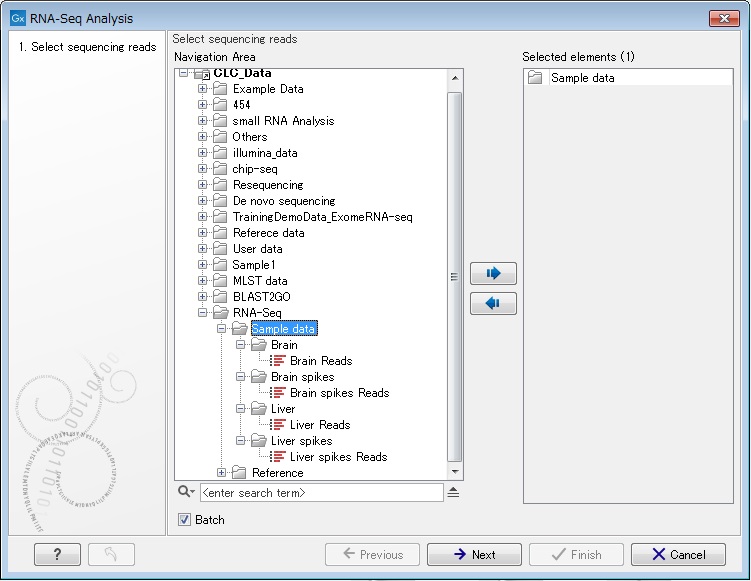
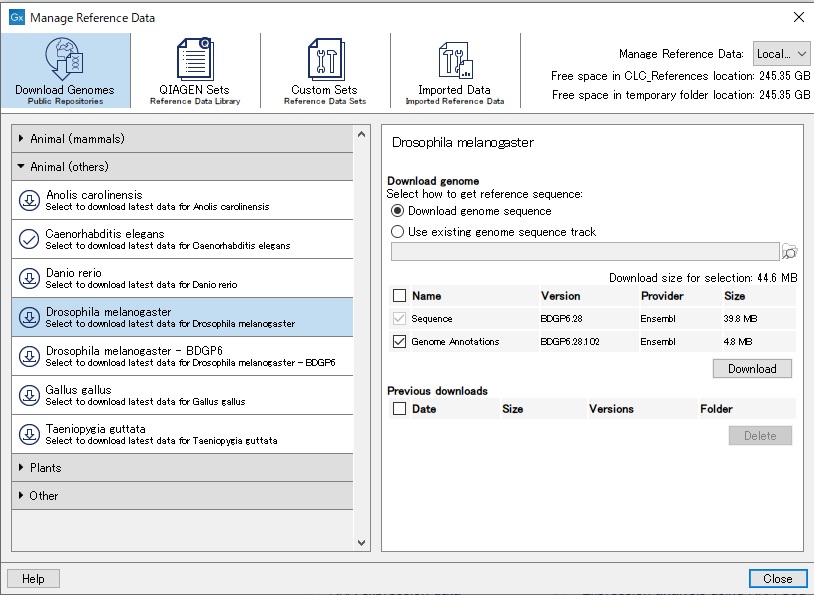
・ゲノムデータのダウンロード
搭載のゲノムデータダウンロードツールを使用すると、主にモデル生物のリファレンスゲノム配列データを容易に取得することができます。また、NCBI GenBank専用のダウンロードツールも搭載し、アクセッション番号やキーワードを入力することで、NCBIに登録されているゲノムデータや配列データをダウンロードすることも可能です。 |
|
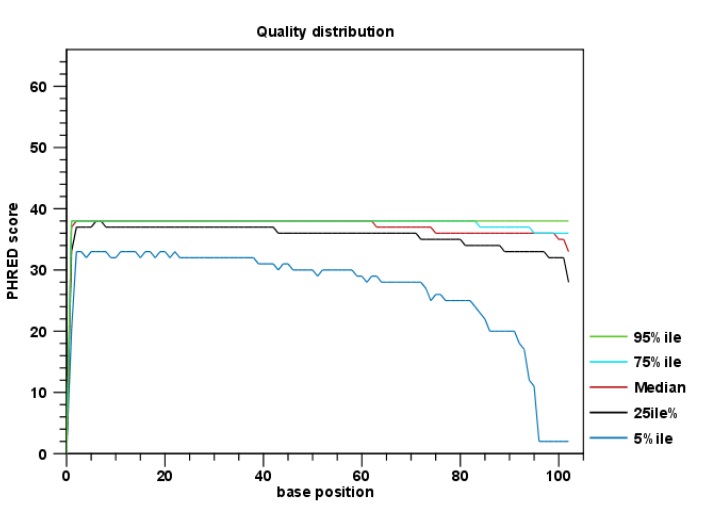
・リード配列データのトリミング/クオリティーチェック
インポートしたリード配列データに対して、配列内の低クオリティー領域やアダプター領域のトリミングを行い、各種データ解析に使用するためのクリーンデータを作成することができます。また、クオリティーチェック用のツールも搭載しており、各種クオリティーチェック項目をまとめたレポートを作成することが可能です。 |
|
(クリックして拡大) |
|
(クリックして拡大) |
|
 サポートするファイルフォーマット サポートするファイルフォーマット

|
| illumina社シークエンサー |
.fastq/.fq |
| Thermo Fisher Scientific社シークエンサー(Ion Torrent) |
.fastq/.fq/.sff/.bam/.sam |
| Pacific Biosciences社シークエンサー |
.bas.h5/.bax.h5/.fastq/.fq/.fasta/.bam/.sam |
| MGI社シークエンサー |
.fastq/.fq |
| Element Biosciences社シークエンサー |
.fastq/.fq |
| Singular Genomics社シークエンサー |
.fastq/.fq |
| Ultima Genomics社シークエンサー |
.cram |
Oxford Nanopore Technologies社シークエンサー
(無償プラグインが必要) |
.fastq/.fq |
| サンガーシークエンサー |
.ab1/.abi/.scf/.phd/.ace |
| その他 |
.fasta/.sam/.bam/.genbank/.gff/.gtf/.bed/.vcf など |
|
 CLC Genomics Workbench Premium CLC Genomics Workbench Premium

 トライアル版ライセンス、メーカー資料の取得 トライアル版ライセンス、メーカー資料の取得

 バージョンアップ履歴、システム要件 バージョンアップ履歴、システム要件

|
下記サイトより、本ソフトウェアのバージョンアップ履歴やシステム要件を確認できます。
|
 関連商品 関連商品

 価格 価格

|
| 商品名 |
ライセンスタイプ |
税別価格 |
Cat.# |
| CLC Genomics Workbench |
Named User License/アカデミック |
¥310,000 |
F-CLC-GW1Y |
| Named User License/コマーシャル |
¥460,000 |
F-CLC-GW1Y-C |
| Concurrent License/アカデミック |
¥460,000 |
F-CLC-GWN1Y |
| Concurrent License/コマーシャル |
¥750,000 |
F-CLC-GWN1Y-C |
CLC Genomics Workbench
Premium |
Named User License/アカデミック |
¥660,000 |
F-CLC-GP1Y |
| Named User License/コマーシャル |
¥910,000 |
F-CLC-GP1Y-C |
| Concurrent License/アカデミック |
¥910,000 |
F-CLC-GPN1Y |
| Concurrent License/コマーシャル |
¥1,290,000 |
F-CLC-GPN1Y-C |
CLC Genomics Workbench
現地取扱説明 |
1回(所要時間:4時間程度) |
お問合せ |
お問合せ |
CLC Genomics Workbench
解析システム |
ご指定のCLC Genomics Workbenchライセンスと解析PCおよびインストール作業を含む |
お問合せ |
クリニカルシークエンス
解析システム |
CLC Genomics Workbench と VarSeq®の各ライセンスと、解析PC及びインストール作業・初期設定作業を含む |
お問合せ |
| ⇒ライセンス形態変更のお知らせ(PDF) |
| * |
テクニカルサポートは、電話あるいは電子メールで対応いたします。 |
| * |
年間ライセンスです。 |
| * |
使用にはインターネット接続が必要です。 |
| * |
Named User License は、名義者のみ利用可能です。また、3台の端末に認証可能ですが、同時に使用できるのは1台です。 |
| * |
Concurrent License のユーザー数に上限はありません。ただし、ユーザーは名義者と同じドメインのメールアドレスをお持ちの方に限ります。また、認証可能な端末数に上限はありませんが、同時に使用できるのは1ライセンスにつき1台です。 |
|
 インテグレーションサービス&充実したサポート体制 インテグレーションサービス&充実したサポート体制

|
【フィルジェンのインテグレーションサービス】
| |
次世代シークエンサーから出力されるデータは膨大であるため、解析を行うコンピューターもそれに対応したスペックのものが必要となります。
フィルジェンでは、CLC Genomics Workbench の一般的なデータ解析に対応したスペックを備えたコンピュータに、CLC Genomics
Workbench をプレインストールしたパッケージの販売を行っております。
詳細は、弊社までお問合せください。 |
 |
【安心・充実のサポート体制】
|
 |
ソフトウェアの価格には、購入後1年間のテクニカルサポートとアップグレードの費用が含まれております。この期間中は、ソフトウェアに関するご質問をメールまたはお電話にて承ります。
さらに、ソフトウェアをよりご活用していただくために、お客様のコンピューターを使用して、「CLC Genomics Workbench」の現地取扱説明会を有償で実施しております。
ご希望の場合は、弊社までお問い合わせください。
|
【クリニカルシークエンス解析システム】
|
CLC Genomics Workbenchとクリニカルシークエンス解析ソフトウェア「VarSeq®」を組み合わせることによって、クリニカルシークエンス分野における、高度なバイオインフォマティクスソリューションを提供いたします。
| ◆ |
データ解析用コンピュータ(Windows OS)が付属し、ご自身でハードウェアをご準備頂く必要がありません。 |
| ◆ |
Clinvarなどの臨床データベースやdbSNP, gnomADなどのアレル頻度データベース、がんゲノム用の知識データベースなどが使用可能です。 |
| ◆ |
全ての解析は、外部サーバーにデータを送信せず、ローカルコンピュータで処理いたしますので、セキュリティーを保つことが可能です。 |
|
|
 |
|
 技術資料・セミナー資料 技術資料・セミナー資料

|
◆技術資料
| CLC Genomics Workbench Premium を利用した ASV 解析と PERMANOVA 解析 |
 |
| CLC Genomics Workbench Premium を使用したメタトランスクリプトーム解析 |
 |
◆セミナー資料
フィルジェン Webセミナー(2026年2月18日)
・CLC Genomics Workbench を用いたリシークエンス解析 |
 |
フィルジェン Webセミナー(2025年10月2日)
・全ゲノム・全エクソーム・ターゲットシークエンスのバイオインフォマティクス解析 |
 |
フィルジェン Webセミナー(2025年7月30日)
・高度なワークフローを用いた複雑なNGSデータ処理プロセスの自動化 |
 |
フィルジェン Webセミナー(2024年12月12日)
・新規転写産物の同定・融合遺伝子の検出 |
 |
フィルジェン Webセミナー(2024年9月10日)
・ロングリードデータを使用した菌叢解析 |
 |
フィルジェン Webセミナー(2023年7月20日)
・microRNAの発現解析(定量解析・アノテーション付与・エンリッチメント解析) |
 |
フィルジェン Webセミナー(2019年8月21日)
・NGSデータ解析入門WEBセミナー:バイサルファイトシークエンス編 |
 |
フィルジェン Webセミナー(2019年3月8日)
・CLC Genomics Workbenchを用いた分子系統樹解析 |
 |
フィルジェン Webセミナー(2018年11月26日)
・NGSデータ解析入門Webセミナー:RNA-Seq解析編 |
 |
フィルジェン Webセミナー(2018年9月28日)
・NGSデータ解析入門Webセミナー:De Novo シークエンス解析編 |
 |
|
|