製品・サービス情報
|
|
ArrayStar社 遺伝子発現制御機構解析
non-coding RNA マイクロアレイ受託解析サービス |
|
 |
 なぜ Downstream-of-Gene Transcripts (DoG RNAs) を研究するのか? なぜ Downstream-of-Gene Transcripts (DoG RNAs) を研究するのか?
|
・DoG RNA とは?
・DoG RNA は疾患における遺伝子転写を調節する
・がん・疾患における DoG 由来キメラ RNA と circular RNA
・DoG RNA のエピジェネティックおよびエピトランスクリプトーム制御
・DoG RNA 研究の道筋
◆DoG RNA とは?
|
Downstream-of-gene (DoG) RNA (遺伝子下流 RNA) は、ホスト遺伝子の~10%の下流で発生し、上流の RNA と連続する
RNA 転写産物です[1]。DoG は、転写終結店(TES)のポリアデニル化シグナル(PAS)を越えて 5kb 以上の長さを持つと定義され、哺乳類細胞では最大
200kb の長さを持ちます[1]。DoG はその長さから、リードイン遺伝子と呼ばれる他の下流転写ユニットとしばしば重複します(図1)[2,
3]。
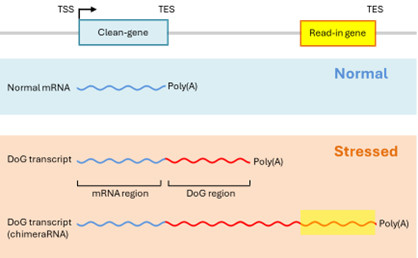
図1.通常の条件下では、リードイン転写のない正常な mRNA を生成。細胞ストレスは「Downstream-of-Gene」転写産物(DoGs)の産生を誘発する。DoG
の中には、「リードイン」遺伝子と呼ばれる、その下流の遺伝子にまで転写を拡大するものがある[2, 3]。合成後、多くの DoG は転写部位の近くに留まるが、一部
DoG は核小胞内に放出される[1, 4]。ポリアデニル化された DoG もポリアデニル化されていない DoG も、同程度のレベルで検出されている[5]。略語:
TSS:転写開始点; TES:転写終結点。
<DoG の分子的特徴>
|
DoG は、リードスルー転写によって発生します。このプロセスは、新生 mRNA の3'末端の終結が効率的でない場合に起こり、遺伝子の正常末端を超えて転写が継続します[6]。DoG
には 3つの特徴があります(図1)[7]:(1)タンパク質をコードするホスト遺伝子のプロモーターから転写が開始される。DoG の産生には、独立した転写開始部位(TSS)からの
de novo 転写開始は含まれない。(2)ホスト遺伝子の 3'末端ポリアデニル化シグナル(PAS)を超えて、少なくとも 5kb にわたって連続的に伸長する。(3)主に核に局在し、クロマチンに結合している可能性が高い。注目すべきは、DoG
の産生はホスト遺伝子の転写活性には依存せず、その調節メカニズムは明確に独立し、関連していないことです。 |
<DoG の生合成>
|
正常な転写終結には、RNAポリメラーゼ RNAPII の減速、一時停止、脱離、RNA転写産物の 3'末端切断など、複数の協調的なイベントが必要です。これらのステップのいずれかが乱れると、転写終結に影響を与え、転写のリードスルーにつながる可能性があります(図2)。浸透圧ストレス[5]、ヒートショック[8]、ウイルス感染[3、9]、がん誘発性突然変異などの細胞ストレスはすべて、転写終結の異常を促進する可能性があります。ストレス条件下では、多くの遺伝子がポリアデニル化シグナル(PAS)を認識できなかったり、転写物の切断やポリアデニル化因子の機能不全により、転写を適切に終結できず、リードスルー転写を促進します。さらに
DoG を産生する遺伝子は、転写伸長を促進するヒストンマーク(例:H3K36me3 や H3K79me2)が濃縮されていたり[10]、PASが弱いなど、しばしばリードスルー転写を起こしやすい特徴的なクロマチン特性を有しています[3]。
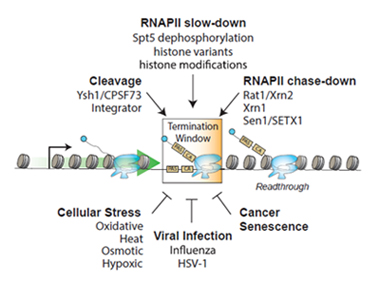
図2.RNAPII 転写終結を促進(上)または阻害(下)する因子、機構、または細胞条件による転写終結の制御。細胞ストレス、ウイルス感染、癌、老化によって引き起こされる転写終結欠陥は、リードスルー転写と
DoG 産生をもたらす可能性がある。
|
【Reference】
| 1. |
Vilborg A et al: Widespread Inducible Transcription
Downstream of Human Genes. Mol Cell 2015, 59(3):449-461.[PMID: 26190259] |
| 2. |
Grosso AR et al: Pervasive
transcription read-through promotes aberrant expression of oncogenes and RNA
chimeras in renal carcinoma. Elife 2015, 4.[PMID: 26575290] |
| 3. |
Rutkowski AJ et al: Widespread
disruption of host transcription termination in HSV-1 infection. Nat Commun
2015, 6:7126.[PMID: 25989971] |
| 4. |
Hennig T et al: HSV-1-induced
disruption of transcription termination resembles a cellular stress response
but selectively increases chromatin accessibility downstream of genes. PLoS
Pathog 2018, 14(3):e1006954.[PMID: 29579120] |
| 5. |
Rosa-Mercado NA et al: Hyperosmotic
stress alters the RNA polymerase II interactome and induces readthrough
transcription despite widespread transcriptional repression. Mol Cell 2021,
81(3):502-513 e504.[PMID: 33400923] |
| 6. |
Rosa-Mercado NA, Steitz JA: Who let the DoGs out? -
biogenesis of stress-induced readthrough transcripts. Trends Biochem Sci 2022,
47(3):206-217.[PMID: 34489151] |
| 7. |
Morgan M, Shiekhattar R, Shilatifard A, Lauberth
SM: It's a DoG-eat-DoG world-altered transcriptional mechanisms drive
downstream-of-gene (DoG) transcript production. Mol Cell 2022,
82(11):1981-1991.[PMID: 35487209] |
| 8. |
Cugusi S et al: Heat shock induces
premature transcript termination and reconfigures the human transcriptome. Mol
Cell 2022, 82(8):1573-1588 e1510.[PMID: 35114099] |
| 9. |
Heinz S et al: Transcription Elongation
Can Affect Genome 3D Structure. Cell 2018, 174(6):1522-1536 e1522.[PMID:
30146161] |
| 10. |
Vilborg A et al: Comparative analysis
reveals genomic features of stress-induced transcriptional readthrough. Proc
Natl Acad Sci U S A 2017, 114(40):E8362-E8371.[PMID: 28928151] |
|
◆DoG RNA は疾患における遺伝子転写を調節する
|
リードスルー転写と DoG RNA の機能的役割は、現在活発な研究の焦点となっており、遺伝子制御への関与を強調するいくつかのメカニズムが提案されています。
・DoG RNA は細胞老化における転写干渉を媒介する
|
DoG はアンチセンス RNA として機能し、収束タンパク質コーディング遺伝子の遺伝子発現を制御します。老化細胞では、老化誘発アンチセンスリードスルー
RNA (senescence-triggered antisense read-through RNAs:START RNA) ファミリーが
DoG として誘導されます[1]。重要な点は、START RNA は対応するセンス RNA の発現を抑制することです。
もう1つは、シロイヌナズナゲノムの低温感受性領域に位置する SVK です。SVK の RNAPII リードスルー転写は、アンチセンス鎖上の CBF1
と重複する不可解な lncRNA-asCBF1 をもたらします[2]。CBF1 は、SVK-asCBF1 lncRNA カスケードに起因する
RNAPII 衝突によって抑制されます(図1)。SVK-asCBF1 カスケードは、低温ストレス時の CBF1 の発現とタイミングを厳密に制御するメカニズムを提供します。
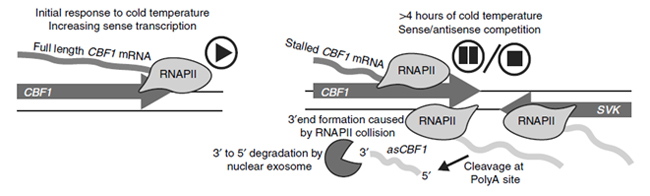
図1.アンチセンス DoG RNA による CBF1 コーディング遺伝子発現の制御
(左)常温では、CBF1 は支障なく転写される。(右)低温曝露により、CBF1 のアンチセンス方向の SVF 転写が増加する。SVK のリードスルー転写により、CBF1
の 3'末端へのアンチセンス転写が起こる。両鎖の転写 RNAPII の占有率が増加すると、RNAPII の衝突が生じ、CBF1 センス転写が停止し、完全長の
CBF1 mRNA 生成する[2]。 |
・DoG RNA はウイルス感染におけるゲノム 3D 構造を制御する
|
A 型インフルエンザウイルス(IAV)感染時、ウイルス非構造タンパク質 NS1 は、高活性ホスト遺伝子のリードスルー転写を誘導し、ホストのクロマチンからのコヒーシン解離、クロマチンループの喪失、リードスルー領域におけるクロマチンの圧縮解除を引き起こします(図2)[3]。
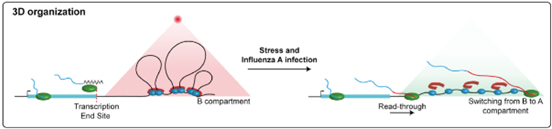
図2.リードスルー転写はクロマチン構造を変化させる
A 型インフルエンザウイルス(IAV)タンパク質 NS1 は転写終結を阻害し、RNAPII が終結部位を通過して進行することを可能にし、クロマチンループの喪失と局所的なクロマチン圧縮解除を引き起こす[3]。 |
【Reference】
| 1. |
Muniz L et al: Control of Gene
Expression in Senescence through Transcriptional Read-Through of Convergent
Protein-Coding Genes. Cell Rep 2017, 21(9):2433-2446.[PMID: 29186682] |
| 2. |
Kindgren P, Ard R, Ivanov M, Marquardt S:
Transcriptional read-through of the long non-coding RNA SVALKA governs plant
cold acclimation. Nat Commun 2018, 9(1):4561.[PMID: 30385760] |
| 3. |
Heinz S et al: Transcription Elongation
Can Affect Genome 3D Structure. Cell 2018, 174(6):1522-1536 e1522.[PMID:
30146161] |
|
◆がん・疾患における DoG 由来キメラ RNA と circular RNA
|
・DoG 由来キメラ RNA
・DoG 由来の circular RNA
|
<リードスルー転写を利用して、下流領域から circular RNA を生成可能>
|
circular RNA は、リードスルー転写から効率的に生成できます。 両側の逆位反復配列が転写されると、バックスプライシングが起こり、RNA
サークルが物理的に形成され、新生転写産物から分離します(図4)[9]。例えば、MATR3-PAIP2 リードスルー転写は、下流の遺伝子 PAIP2
から circPAIP2 を産生します (図4)[9]。
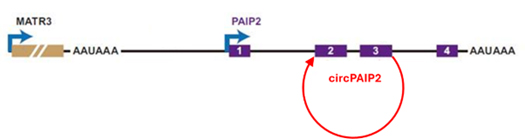
図4.MATR3-PAIP2 のリードスルー転写により、下流の PAIP2 のエクソン 3 から 2 へバックスプライシングされ、circPAIP2
が生成される[9]。 |
<リードスルー circular RNA (rt-circRNA)>
|
最近、リードスルー circRNA(rt-circRNA) と呼ばれる新しいタイプの circRNA が発見されました (図5)。これらは隣接する
2つの同様の方向にある遺伝子からのエクソンをコードするハイブリッド circular RNA です。Vo らは 1,359 個のヒト rt-circRNA
を報告しました[10]。
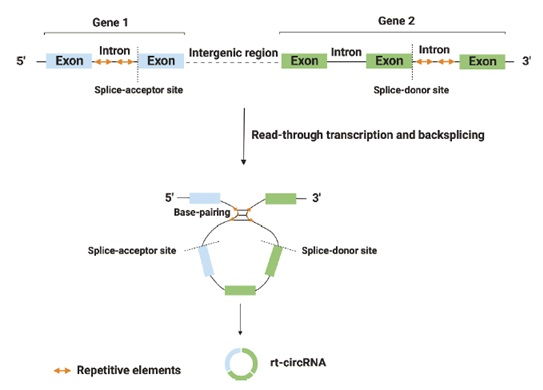
図5.rt-circRNA の生合成
リードスルー転写により、2つの隣接し、同様の方向を向いた 2つの遺伝子からコーディングエクソンを含むハイブリッド circRNA (rt-circRNA) が形成される。環状化プロセスは、反復配列を持つ長いイントロン間の塩基対形成によって媒介される [11]。
|
|
【Reference】
| 1. |
Shi X, Singh S, Lin E, Li H: Chimeric RNAs in
cancer. Adv Clin Chem 2021, 100:1-35.[PMID: 33453863] |
| 2. |
Qin F et al: Discovery of
CTCF-sensitive Cis-spliced fusion RNAs between adjacent genes in human prostate
cells. PLoS Genet 2015, 11(2):e1005001.[PMID: 25658338] |
| 3. |
Rickman DS et al: SLC45A3-ELK4 is a
novel and frequent erythroblast transformation-specific fusion transcript in
prostate cancer. Cancer Res 2009, 69(7):2734-2738.[PMID: 19293179] |
| 4. |
Zhu D et al: The landscape of
chimeric RNAs in bladder urothelial carcinoma. Int J Biochem Cell Biol 2019,
110:50-58.[PMID: 30818082] |
| 5. |
Zhang Y et al: Chimeric transcript
generated by cis-splicing of adjacent genes regulates prostate cancer cell
proliferation. Cancer Discov 2012, 2(7):598-607.[PMID: 22719019] |
| 6. |
Kumar-Sinha C, Kalyana-Sundaram S, Chinnaiyan AM:
SLC45A3-ELK4 chimera in prostate cancer: spotlight on cis-splicing. Cancer
Discov 2012, 2(7):582-585.[PMID: 22787087] |
| 7. |
Qin F, Zhang Y, Liu J, Li H: SLC45A3-ELK4 functions
as a long non-coding chimeric RNA. Cancer Lett 2017, 404:53-61.[PMID: 28716526] |
| 8. |
Grosso AR et al: Pervasive
transcription read-through promotes aberrant expression of oncogenes and RNA
chimeras in renal carcinoma. Elife 2015, 4.[PMID: 26575290] |
| 9. |
Liang D et al: The Output of
Protein-Coding Genes Shifts to Circular RNAs When the Pre-mRNA Processing
Machinery Is Limiting. Mol Cell 2017, 68(5):940-954 e943.[PMID: 29174924] |
| 10. |
Vo JN et al: The Landscape of Circular
RNA in Cancer. Cell 2019, 176(4):869-881 e813.[PMID: 30735636] |
| 11. |
Vidal AF: Read-through circular RNAs reveal the
plasticity of RNA processing mechanisms in human cells. RNA Biol 2020,
17(12):1823-1826.[PMID: 32783578] |
|
◆DoG RNA のエピジェネティックおよびエピトランスクリプトーム制御
|
・H3K36 トリメチル化の調節異常
|
明細胞腎細胞癌 (ccRCC) では、転写終結の欠陥とそれに伴うスプライシングの異常により、リードスルー転写と DoG 形成が広範囲に及んでいます [1]。特に、H3K36 メチルトランスフェラーゼ遺伝子 SETD2 は頻繁に変異しています。ccRCC 細胞の SETD2 ノックアウトは、リードスルー転写を誘導しました [1]。リードスルー転写はさまざまな癌でも観察されており、この転写欠陥が細胞形質転換の一般的な原因であることを裏付けています [2, 3]。
|
・ヒストン H2A.Z 欠失
|
ヒストン H2A.Z は、DNA 修復に関連する代替ヒストン変異体です。DoG は、H2A.Z が存在しない非増殖性老化細胞で検出されます (図1)[4]。DoGは、アンチセンス転写産物として作用することにより、遺伝子発現を制御する上で重要な役割を果たすと考えられています。
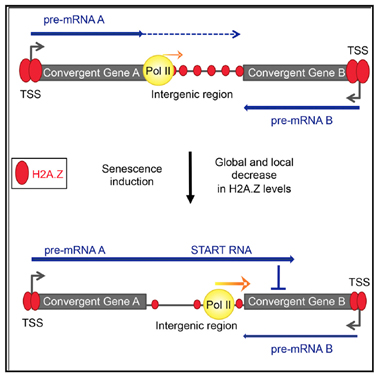
図1.収束性タンパク質コーディング遺伝子の転写リードスルーによる老化における遺伝子発現の制御
老化の過程では、START RNA と呼ばれる機能性アンチセンス RNA ファミリーが、収束遺伝子の下流の転写リードスルーによって生成される。これらの
RNA は、POLII の伸長速度と H2A.Z の局所占有率の制御に依存するメカニズムによって活性化される[4]。 |
・RNA 修飾
|
m6A などの RNA 修飾は、転写終結に影響を与えます [5, 6]。植物では、m6A ライター関連因子 FIP37 の変異体が、遺伝子のサブセットでリードスルー転写とキメラ mRNA の形成を誘導します [7]。m6A 支援ポリアデニル化 (m6A–assisted
polyadenylation: m-ASP) 経路はトランスクリプトームの完全性を確保しますが、これには m6A ライター関連因子 FIP37 および m6A リーダー CPSF30L が必要です [7]。FIP37 および CPSF30L 欠損植物における標的 m-ASP 経路は、転写リードスルーと mRNA キメラ形成を引き起こします (図2)。さらに、m-ASP 経路は、キメラ遺伝子/転移因子転写産物の形成を制限し、特定の遺伝子座における転移因子を制御する可能性もあります。まとめると、3'-UTR m6A の選択的認識は、不適切な遺伝子発現を制限し、トランスクリプトームの完全性を確保するためのセーフガード機構として機能します [7]。
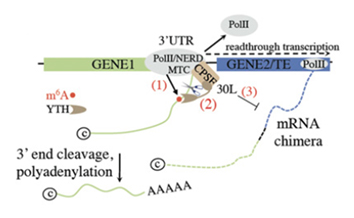
図2.DoG 形成における m6A 支援ポリアデニル化(m-ASP)経路のモデル[7]
(1) GENE1 の 3'-UTR における m6A 導入には、m6A ライター関連因子 FIP37 が必要である。(2)m6A は CPSF30L リーダーの YTHDC 型ドメインに認識され、GENE1 の 3'-UTR での切断とポリアデニル化を促進される。(3)これにより、mRNA キメラの形成が制限される。
|
・m6A 修飾と R-loop
|
新生 RNA m6A 修飾は、遺伝子の転写終結領域における R-loop 形成を推進し、転写終結を促進します(図3)[8]。m6A メチルトランスフェラーゼ METTL3 の枯渇により、TES 周辺の m6A 遺伝子における R-loop の蓄積が劇的に減少し、その結果、転写終結欠陥とリードスルー転写を引き起こします [8]。影響を受けた TES での R-loop の回復と、それに伴うリードスルー活性の抑制には、METTL3 メチルトランスフェラーゼ活性が必要です [8]。
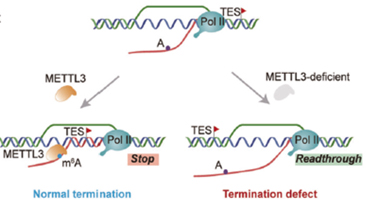
図3.m6A 修飾は、転写終結領域における R-loop 形成を推進し、効率的な転写終結を促進する[8]
最近、R ループは DDX21 と METTL3 のリクルートのためのクロマチンアンカーとして機能し、これらが一緒になって新生転写産物への m6A
の共転写を促進することが発見されました。新生 RNA 上の m6A の存在は、主要な核内の 5'-3'エキソリボヌクレアーゼである XRN2
のリクルートとローディングに重要な役割を果たしています。転写終結の魚雷モデル(torpedo model)では、XRN2 は poly(A)
切断部位に侵入し、進行中の転写から生じた RNA を分解し、RNAPII を追いかけて DNA から除去し、効果的に転写を終結させます。YTHDC1
などの m6A リーダータンパク質は、転写終結部位に XRN2 を結合してリクルートし、終結を促進することが可能です。DDX21、METTL3、またはそれらの酵素活性の欠失など、これらのステップのいずれかが混乱すると、不完全な終結と転写リードスルーにつながります(図4)[9]。
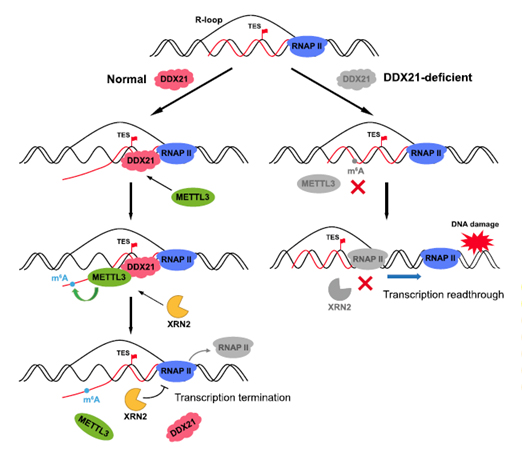
図4.DDX21-METTL3-m6A 系は、新生 RNA に m6A を導入し、TES の R-loop を解消し、XRN2 をリクルートして転写を終了させる。これらの活性の欠失は、転写終結の欠陥、DoG
形成、およびそれに伴う DNA 損傷につながる可能性がある[9]。 |
【Reference】
| 1. |
Grosso AR et al: Pervasive transcription
read-through promotes aberrant expression of oncogenes and RNA chimeras in
renal carcinoma. Elife 2015, 4.[PMID: 26575290] |
| 2. |
Maher CA et al: Transcriptome sequencing to detect
gene fusions in cancer. Nature 2009, 458(7234):97-101.[PMID: 19136943] |
| 3. |
Kannan K et al: Recurrent chimeric RNAs enriched in
human prostate cancer identified by deep sequencing. Proc Natl Acad Sci U S A
2011, 108(22):9172-9177.[PMID: 21571633] |
| 4. |
Muniz L et al: Control of Gene Expression in
Senescence through Transcriptional Read-Through of Convergent Protein-Coding
Genes. Cell Rep 2017, 21(9):2433-2446.[PMID: 29186682] |
| 5. |
Frye M, Harada BT, Behm M, He C: RNA modifications
modulate gene expression during development. Science 2018,
361(6409):1346-1349.[PMID: 30262497] |
| 6. |
Anreiter I et al: New Twists in Detecting mRNA
Modification Dynamics. Trends Biotechnol 2021, 39(1):72-89.[PMID: 32620324] |
| 7. |
Pontier D et al: The m(6)A pathway protects the
transcriptome integrity by restricting RNA chimera formation in plants. Life
Sci Alliance 2019, 2(3).[PMID: 31142640] |
| 8. |
Yang X et al: m(6)A promotes R-loop formation to
facilitate transcription termination. Cell Res 2019, 29(12):1035-1038.[PMID:
31606733] |
| 9. |
Hao JD et al: DDX21 mediates co-transcriptional RNA
m(6)A modification to promote transcription termination and genome stability.
Mol Cell 2024, 84(9):1711-1726 e1711.[PMID: 38569554] |
|
◆DoG RNA 研究の道筋
|
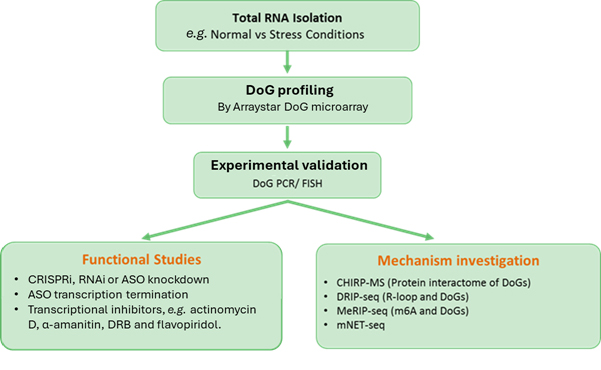
図5.DoG RNA 研究の道筋
・発現変動の検証
|
アレイプロファイリングで差次的に発現した DoG RNA は、発現レベルの独立した qPCR 定量によって確認することができます。さらに、RNA
蛍光 in situ ハイブリダイゼーション(FISH)により、DoG RNA の強度と DoG RNA の細胞内分布の両方に関する情報を得ることができます。 |
・細胞内分画と局在化
|
DoG
RNA は、核内クロマチン画分に強く濃縮されていることが知られており、細胞質内の同族 mRNA とは区別されています。細胞質、可溶性核、およびクロマチン細胞内画分から単離された Total
RNA の qPCR 解析は、DoG RNA 細胞内分布と機能的意味を理解するのに役立ちます。 |
・CRISPR 干渉
|
CRISPR 干渉(CRISPR interference:CRISPRi)は、上流遺伝子の転写をノックダウンするために用いることができ、DoG
RNA 産生への影響や DoG RNA との関係を調べるのに役立ちます。CRISPRi では、触媒的に死滅した Cas9 (dCas9)が標的遺伝子に誘導され、切断することなくその発現を阻害します。dCas9
ドメインは、RNA ポリメラーゼ II(Pol II)の立体阻害を通じてサイレンシングを最大化するために、しばしば抑制エフェクター(例えば KRAB
ドメインなど)と融合します。
以下は、SRSF1 遺伝子のプロモーターに CRISPRi を使用して、ホスト遺伝子とその DoG RNA の両方の発現をノックダウンした例です(図6)。

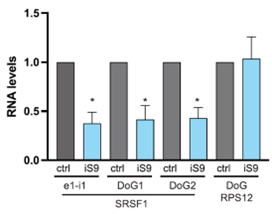
図6.SRSF1 遺伝子転写の CRISPRi ノックダウン(上)は、SRSF1 pre-mRNA と DoG RNA の両方を、qPCR で定量化(下)したのと同じ 40% のレベルまで協調的に減少さる。Ctrl
:CRISPRi を含まないネガティブコントロール細胞; iS9:CRISPRi ノックダウン細胞。 e1–i1 : SRSF1 pre-mRNA 領域; DoG1およびDoG2: SRSF1 DoG 領域。 |
・アンチセンスオリゴ(Antisense Oligo: ASO)
|
ホスト遺伝子 mRNA 発現の ASO ノックダウンは、対応する DoG RNA のレベルを低下させることができます。重要なことに、ASO は早期の転写終結を引き起こすことも知られており、これは DoG ホスト遺伝子の任意の領域を標的にして、転写終結が DoG RNA どのように生成するかを観察するのに特に有効です [23, 24]。ASO (ここでは gapmer も)による転写終結は、内在性 RNase H による標的 RNA 切断によって媒介され、正常な転写終結の魚雷モデルと同様にエキソヌクレアーゼ XRN2 を必要とします[24, 25]。
|
・DRIP-seq
|
R-loops は、遺伝子プロモーターや転写終結領域に多く存在し、どちらも DoG ホスト遺伝子の転写や DoG RNA 形成にかかわる転写終結に重要な役割を果たしています。DRIP-seq とその類似解析は、R ループプロファイリングの一般的な方法であり、R-loop と DoG の制御に関する貴重な洞察を提供します。
|
・MeRIP-seq
|
転写終結領域に濃縮された RNA 修飾(例:m6A)は、転写終結に影響を与える可能性があります。転写終結点(TES9 での m6A の減少は、終結欠陥とリードスルー転写をもたらします[25]。MeRIP-seq
は、RNA 修飾と DoG RNA の関連付けに使用できるツールです。 |
・mNET-seq
|
Mammalian
Native Elongating Transcript Sequencing (mNET-seq) は、RNA ポリメラーゼII にまだ結合している新生 RNA 転写産物を捕捉してプロファイリングし、DoG RNA に関連する Pol II の一時停止、共転写 RNA プロセッシング、終結などを含む転写のさまざまな段階の解析を可能にします。
|
・ChIRP-MS
|
質量分析による RNA 結合タンパク質の包括的な同定 (Comprehensive
identification of RNA-binding proteins by mass spectrometry:ChIRP-MS) は、RNA と相互作用するタンパク質をトランスクリプトームワイドに同定するために使用されてきました。DoG RNA にどのようなタンパク質因子が結合しているかを知ることで、DoG RNA の機能やメカニズムを導き出すことができます。
|
・Chimera RNA Analysis
|
キメラ RNA (cis-SAGeとも呼ばれる)は、隣接する遺伝子のシス-スプライシング/リードスルーによって形成される DoG RNA の一種です。siRNA
ノックダウンと qPCR 定量を併用することで、キメラ RNA の発現、機能、分子メカニズムを解剖することができます(図5)[6]。
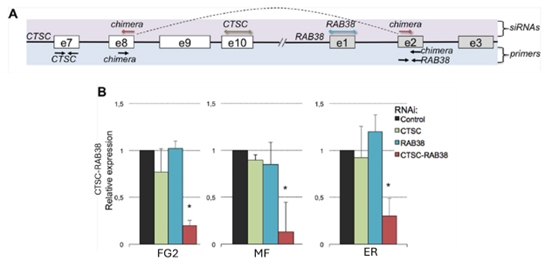
図5.(A)CTSC、RAB38、およびキメラ CTSC-RAB38 転写産物を標的とする siRNA を示す CTSC-RAB38 キメラ
RNA 遺伝子座。これらの転写産物は、qPCR プライマーによって定量された。破線の曲線は、キメラ RNA を作成するスプライシングを表している。(B)3つの異なる
ccRCC 細胞株 FG2、MF、および ER における CTSC。RAB38、および CTSC-RAB38 領域における RNAi ノックダウン後の相対的な
CTSC-RAB38 キメラ RNA 発現。 *コントロールと比較した T 検定により、p<0.05[6]。 |
|
|
*本サイトの情報は、Arraystar社のHomepageの情報を一部引用しております。
|
|