製品・サービス情報
|
|
ArrayStar社 遺伝子発現制御機構解析
non-coding RNA マイクロアレイ受託解析サービス |
|
 |
| Arraystar社の各種アレイを用いた受託解析サービスをご提供いたします。ベストセラーである LncRNA マイクロアレイをはじめ、CircRNA マイクロアレイ、T-UCR マイクロアレイ、piRNA マイクロアレイ、DoG RNA マイクロアレイ、GlycoRNA マイクロアレイの受託サービスを提供しています。 |
 CircRNA Arrays 受託解析サービス CircRNA Arrays 受託解析サービス

|
Circular RNA Microarray は、調節的機能を有すると考えられている新規 long non-coding RNA - circular
RNA の発現を体系的にプロファイルします。Linear RNAに対する相補性、ヌクレアーゼに対する安定性、miRNAによる分解への抵抗性、そして多数の
miRNA 結合部位により、いくつかの circRNA は遺伝子発現において極めて効果的な天然の miRNA スポンジとして認識されており、また、豊富に発現していることが見出されてきています。その細胞特異的発現および長い半減期は、新規バイオマーカーとしての優れた利点を示しています。Arraystar
CircRNA Array は、この新らたに注目されている分野の non-coding RNA を最も効果的にプロファイルおよび解析、探索するためのサービスです。
|
|

カタログダウンロード(PDF) |
|
【特長】
|
◆ |
高感度かつ特異的にcircular RNAをプロファイリングする実用的なプラットフォーム |
| ◆ |
CircRNA発現を確実にプロファイリング: Circular junction-specific プローブの採用、linear RNAの除去
Circular junction-specific プローブの採用により、相対するlinear RNAの存在下でも、個々のcircRNAを確実かつ正確に同定します(図1)。また、RNase
R処理によりlinear RNAを分解することでcircular RNAを回収します。
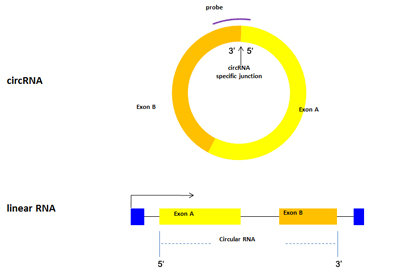 |
図1. Arraystar circRNA Microarray V2.0は、相対するlinear RNA存在下でも各circRNAを正確かつ確実に検出できる様、circular
junction specific probeを使用しています。
下部に示されているlinear RNAは、上部の環状バリアントを産生するために選択的プロセッシングがなされます。プローブは、エクソンAの5'末端とエクソンBの3'末端が結合するcircRNA特異的ジャンクション部位を標的とする様に設計されています。 |
|
|
◆ |
CircRNAに関する詳細なアノテーション: miRNA結合部位やmiRSVRスコア、保存状態など
Circular RNA上にあるmiRNAの潜在的な標的部位に関するアノテーションは、circRNAの有するmiRNAスポンジ機能の解明に貢献します(図2)。
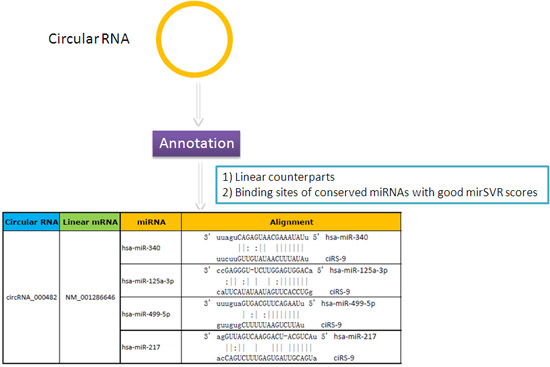
図2. Circular RNAとmiRNA間の関連性は、詳細にアノテーションされます。 |
|
◆ |
RNA-Seqよりも優れたプロファイリング
Circular RNAの以下の特徴から、現在のRNA-Seqよりも好ましい選択肢であると考えられています。
・Circular RNAは、linear RNAと比して発現レベルが低い
・RNA-Seqにおけるcircular junctionのリードは少量
・RNA-Seqによる再現性ある発現変動解析には、数百リードが必要
ArraystarのcircRNAマイクロアレイでは、circular junction-specific probeとRNase処理によるlinear
RNAの除去により、細胞当たりの1転写産物レベルの感度で、circular RNAを特異的に回収します(図4)。これらは現在、実用的かつ成熟したcircular
RNAプロファイリングの技術となっています。
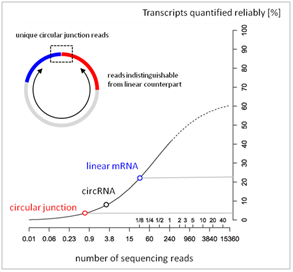
図3.RNA-Seqによる定量の信頼性 vs リード深度
典型的なRNA-SeqのmRNAに対する深度は<300万リードですが(青丸)、この場合のcircular junctionリードは<50万です(赤丸)。確実に定量されるcircular
junctionは5%以下です。
*Adopted from Labaj et al, (2011) Bioinformatics[PMID 21685096]. |
|
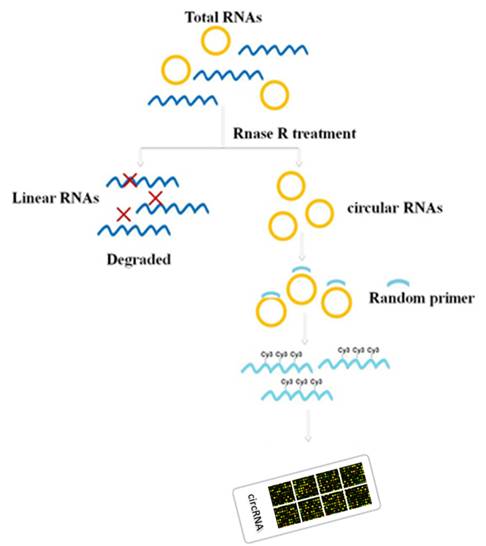
図4.circular junction specific probeとRNase R 前処理による、circular RNA の回収 |
⇒詳細はこちら |
|
◆ |
保証されたパフォーマンス: 広いダイナミックレンジ、高い再現性 |
|
| |
【データベース】
|
◆Human
|
| 総プローブ数 |
13,617 |
| プローブ長 |
60 nt |
| プローブの選択領域 |
circRNA-specific junction を標的とするプローブ |
| プローブの特異性 |
転写物特異的 |
| ラベリング方法 |
ランダムプライマー標識とRNase R サンプル前処理を組み合わせた、CircRNAの特異的かつ効率的な標識 |
| Circular RNA ソース |
Salzman's circRNAs[4]: 8,529
Memczak's circRNAs[3]: 1,601
Zhang's circRNAs[6]: 93
Zhang's circRNAs[5]: 4,980
Jeck's circRNAs[2]: 3,769
Guo's circRNAs[1]: 5,536 |
| アレイフォーマット |
8x15K |
|
◆Mouse
|
| 総プローブ数 |
14,236 |
| プローブ長 |
60 nt |
| プローブの選択領域 |
circRNA-specific junction を標的とするプローブ |
| プローブの特異性 |
転写物特異的 |
| ラベリング方法 |
ランダムプライマー標識とRNase R サンプル前処理を組み合わせた、CircRNAの特異的かつ効率的な標識 |
| Circular RNA ソース |
Memczak's circRNAs: 1,750
Guo's circRNAs[1]: 570
You's circRNAs[7]: 13,300 |
| アレイフォーマット |
8x15K |
|
◆Rat
|
| 総プローブ数 |
14,145 |
| プローブ長 |
60 nt |
| プローブの選択領域 |
circRNA-specific junction を標的とするプローブ |
| プローブの特異性 |
転写物特異的 |
| ラベリング方法 |
ランダムプライマー標識とRNase R サンプル前処理を組み合わせた、CircRNAの特異的かつ効率的な標識 |
| Circular RNA ソース |
You Xintian's circRNAs[7]: 12,298
Mouse circRNA orthologs: 1,668
Human circRNA orthologs: 179 |
| アレイフォーマット |
8x15K |
|
|
【各データベースに関する参考文献】
|
1. |
|
Guo, J. U., V. Agarwal, et al. (2014). "Expand identification and
characterization of mammalian circular RNAs." Genome Biol 15(7): 409. |
| 2. |
|
Jeck, W. R., J. A. Sorrentino, et al. (2013). "Circular RNAs are abundant,
conserved, and associated with ALU repeats." RNA 19(2): 141-157. |
| 3. |
|
Memczak, S., M. Jens, et al. (2013). "Circular RNAs are a large class
of animal RNAs with regulatory potency." Nature 495(7441): 333-338. |
| 4. |
|
Salzman, J., R. E. Chen, et al. (2013). "Cell-type specific features
of circular RNA expression." PloS Genet 9(9): e1003777. |
| 5. |
|
Zhang, X.O., H. B. Wang, et al. (2014). "Complementary sequence-mediated
exon circularization." Cell 159(1): 134-147. |
| 6. |
|
Zhang, Y., X. O. Zhang, et al. (2013). "Circular intronic long noncoding
RNAs." Mol Cell 51(6): 792-806. |
| 7. |
|
You X., I. Vlatkovic, et al. (2015). "Neural circular RNAs are derived
from synaptic genes and regulated by development and plasticity."
Nat Neurosci 18(4): 603-610. |
【ワークフロー】
|
Arraystarは、サンプル調整からデータ解析まで、circular RNA Microarrayプロファイリングのフルサービスを提供します。段階的な品質管理は、信頼できる結果の取得を確かなものとする様にデザインされています。お客様にはサンプルをご提供いただくだけです。サンプルのご準備については、Sample Submission Guideline をご参照ください。
・RNA 品質チェック(QC)
・total RNA の純度と濃度の決定
・total RNA の整合性評価
・RNase R 処理
・cDNA 合成
・ラベリング
・アレイハイブリダイゼーション、洗浄、スキャンニング
・データ抽出、解析、要約
・合理化・最適化されたラベリングパイプライン |
【バイオインフォマティクス解析】
|
◆CircRNA-miRNA関連性の詳細なアノテーション
|
CircRNAには、miRNAの潜在的な標的部位情報がアノテーションされるため、そのmiRNAスポンジとしての機能の解明に有益です。
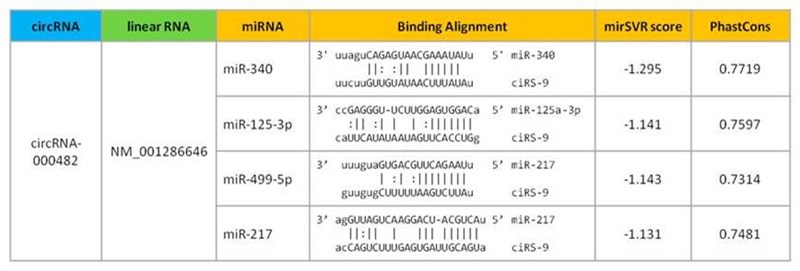
図1. Circular RNAおよびそれと関連するmiRNAの詳細なアノテーション |
◆発現変動circRNA
|
発現変動circRNAは、変化の規模および統計的有意差(p-value)によって選択されます(これは、ボルケノプトットで可視化できます)。発現様式は、階層型クラスター分析ヒートマップによって表示されます。

図2. 発現変動circRNAにおける、複数のmicroRNAとの結合性およびMRE(microRNA Response Element)の詳細 |
|
【参考文献】
【税別価格】
|
| アレイ名 |
生物種 |
内容 |
サンプル数 |
税別単価※ |
Cat.# |
Circular RNA Microarray
受託解析サービス |
ヒト |
13,617 circular RNAs
8×15K |
1サンプル
から受付 |
¥150,000 |
F-AS-CIRH-(サンプル数) |
| マウス |
14,236 circular RNAs
8×15K |
F-AS-CIRM-(サンプル数) |
| ラット |
14,236 circular RNAs
8×15K |
F-AS-CIRR-(サンプル数) |
※ 1サンプルの解析料金です。海外提携先への送料が別途必要となります。 |
|
 解析の流れと必要サンプル量 解析の流れと必要サンプル量

*本サイトの情報は、Arraystar社のHomepageの情報を一部引用しております。
|
|