製品・サービス情報
|
|
Biomarker Technologies社(BMKGene) 次世代シークエンシング(NGS)受託解析サービス
空間トランスクリプトーム受託解析サービス
(10x Genomics Visium) |
|
 |
| 空間トランスクリプトミクスは、研究者が空間的文脈を維持しながら組織内の複雑な遺伝子発現パターンを調査できる最先端の技術です。この技術を使用することで、研究者は細胞の空間的構成と組織内で発生する複雑な分子相互作用についてより深く理解することができ、腫瘍学、神経科学、発生生物学、免疫学、植物学など、複数の分野における生物学的プロセスの根底にあるメカニズムについて貴重な洞察を得ることができます。 |
 10x Genomics Visium 空間トランスクリプトーム 10x Genomics Visium 空間トランスクリプトーム

|
空間トランスクリプトーム解析における強力なプラットフォームの 1 つに、illumina シークエンシングの組み合わせた 10x Genomics
Visium が挙げられます。10x visium の原理は、組織切片が置かれるキャプチャー領域が指定された特殊なチップにあります。このキャプチャー領域には、バーコード化されたスポットがあり、それぞれが組織内の固有の空間位置に対応しています。組織からキャプチャーされた
RNA 分子は、逆転写の過程で、固有の分子識別子(UMA)で標識されます。これらのバーコード化スポットと UMI は、シングルセル分解能で遺伝子発現の正確な空間マッピングと定量化を可能にします。空間的にバーコード化されたサンプルと
UMI の組み合わせにより、生成されるデータの精度と特異性が保証されます。
◆サービスの概要
|
【技術概要】
【特徴】
|
| ・ |
解像度:100μM |
| ・ |
スポット径:55μM |
| ・ |
スポット数:約 4492 個 |
| ・ |
キャプチャー領域:6.5mm×6.5mm |
| ・ |
各バーコードビーズには、以下の 4 つのセクションで構成されたプライマーを搭載:
-mRNA プライミングおよび cDNA 合成のための poly(dT)テール
-増幅バイアスを補正するための Unique Molecular Identifier (UMI)
-空間バーコード
-部分的な Read1 のシークエンシングプライマーの結合配列 |
| ・ |
切片の H&E 染色 |
|
【10x Genomics Visium の特長】
|
・ワンストップサービス:
|
凍結切片作成、染色、組織最適化、空間バーコーディング、ライブラリー調製、シークエンシング、バイオインフォマティクスなど、経験とスキルに基づくすべてのステップを統合します。 |
・高度なスキルを持つ技術チーム:
|
ヒト、マウス、哺乳類、魚類、植物など、250 種類以上の組織タイプと 100 以上の生物種の経験があります。 |
・プロジェクト全体のリアルタイム更新:
・包括的なバイオインフォマティクスとユーザーフレンドリーな結果の視覚化:
|
解析サービスパッケージには、29 の分析と 100 を超える高品質の図が含まれています。 |
・カスタマイズデータ解析と視覚化:
|
【サービスの仕様】
|
| 必要サンプル条件 |
QCT 包埋クライオサンプル(最適径:約 6×6×6 mm3)
サンプルあたり 3 ブロック |
| ライブラリー |
10x Visium cDNA ライブラリー |
| シークエンス条件 |
illumina PE150 |
| 推奨データ量 |
スポットあたり 50K PE reads (60Gb) |
| サンプルQC |
RIN>7 |
|
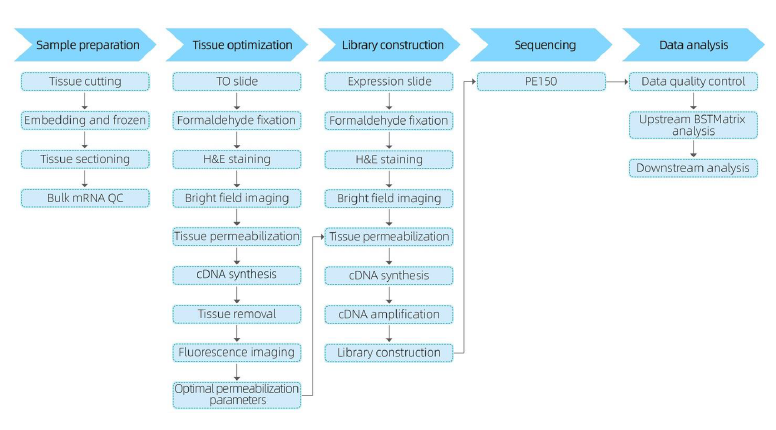
【ワークフロー】
|
サンプル調製段階では、高品質の RNA を確実に得られる様、最初のバルク RNA 抽出試験が行われます。組織最適化段階では、切片を染色して可視化し、組織から
mRNA を放出するための透過条件も最適化します。最適化されたプロトコールによりライブラリーを作製し、その後、シークエンシングとデータ解析が行われます。
 |
|
◆バイオインフォマティクス解析
|
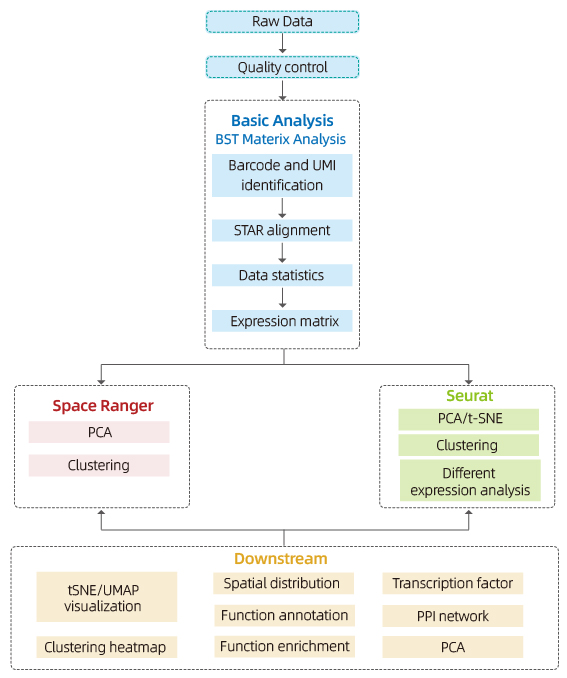
本サービスには、以下の解析が含まれています。
・データ品質管理
データ出力と品質スコアの分布
スポットあたりの遺伝子検出
組織カバー率
・サンプル内分析
遺伝子の豊富さ
スポットクラスタリング(次元削減分析を含む)
クラスター間の発現差解析:マーカー遺伝子の同定
マーカー遺伝子の機能アノテーションとエンリッチメント
・グループ間分析
両サンプル(例:疾患とコントロール)由来のスポットを組み合わせて、再クラスター化
各クラスターのマーカー遺伝子の同定
マーカー遺伝子の機能アノテーションとエンリッチメント
同じクラスターのグループ間発現変動 |
◆解析結果例
|
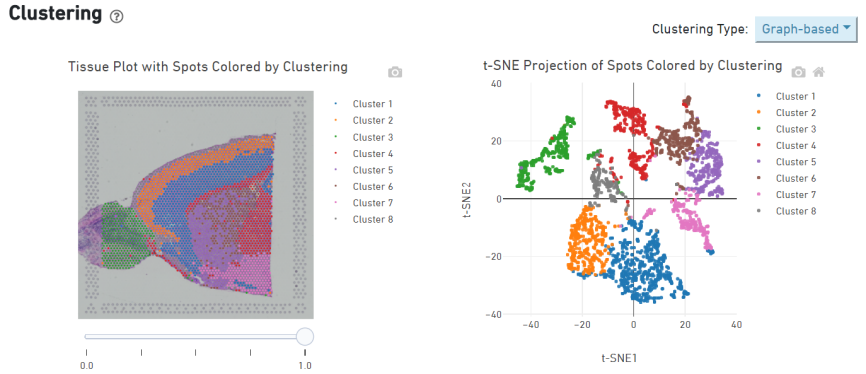
<サンプル内分析>
|
スポットクラスタリング:
マーカー遺伝子の同定と空間分布
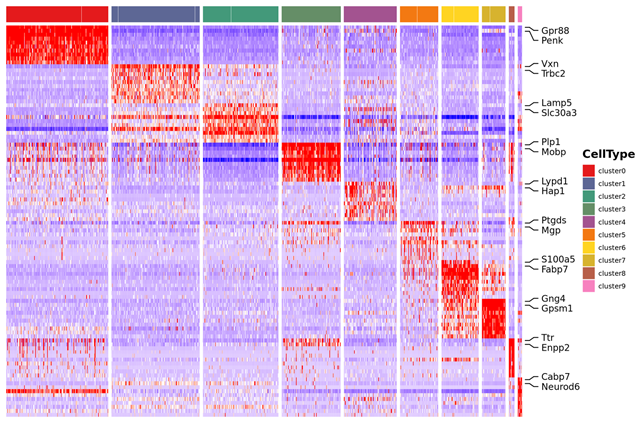
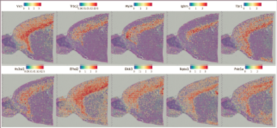
|
<グループ間分析>
|
両グループのデータの組み合わせと再クラスタリング
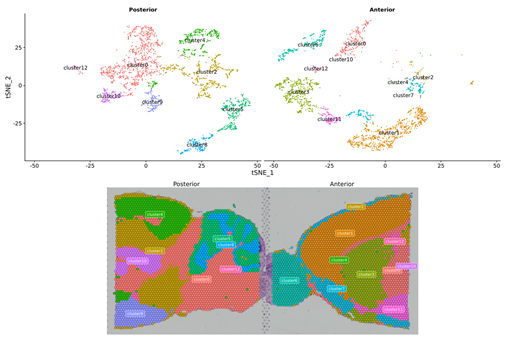
新規クラスターのマーカー遺伝子

|
|
【参考文献】
|
10x Visium による BMKGene の空間トランスクリプトミクスサービスがもたらす進歩を、以下の特集記事でご覧ください。
| ・ |
Chen,
D. et al. (2023) ‘mthl1, a potential Drosophila homologue of mammalian adhesion
GPCRs, is involved in antitumor reactions to injected oncogenic cells in
flies’, Proceedings of the National Academy of Sciences of the United States of
America, 120(30), p. e2303462120. doi: /10.1073/pnas.2303462120
|
| ・ |
Chen,
Y. et al. (2023) ‘STEEL enables high-resolution delineation of spatiotemporal
transcriptomic data’, Briefings in Bioinformatics, 24(2), pp. 1–10. doi:
10.1093/BIB/BBAD068.
|
| ・ |
Liu,
C. et al. (2022) ‘A spatiotemporal atlas of organogenesis in the development of
orchid flowers’, Nucleic Acids Research, 50(17), pp. 9724–9737. doi:
10.1093/NAR/GKAC773.
|
| ・ |
Wang,
J. et al. (2023) ‘Integrating Spatial Transcriptomics and Single-nucleus RNA
Sequencing Reveals the Potential Therapeutic Strategies for Uterine Leiomyoma’,
International Journal of Biological Sciences, 19(8), pp. 2515–2530. doi:
10.7150/IJBS.83510.
|
|
|
 ご注意事項 (必ずご確認ください) ご注意事項 (必ずご確認ください)

|
1. |
解析サービスに関して
本サービスの解析作業は、弊社の海外提携先(Biomarker Technologies(BMKGene)社にて実施されます。 |
| 2. |
お見積りに関して
正式なお見積書は、直接販売の場合には直接ご提出いたしますが、代理店経由の場合は代理店よりご提出いたします。 |
| 3. |
サンプルの送付および取り扱いに関して
| 3.1 |
サンプルのご発送は、必ず、サンプル送付方法およびご注意点(PDF)の内容に従ってください。送付の際は、チューブをパラフィルムで覆い、キャップの緩みや中身の漏れがおきない様にしてください。サンプルの入ったチューブには、油性マジックでサンプル名を明記し、ビニールバッグ等に入れてください。 |
| 3.2 |
サンプルは、なるべく休日をはさまない様に、弊社営業時間内(土日祝日、年末年始等を除く、平日9時~18時)に到着する様にご発送ください。なお、ご発送時の送料はお客様負担となります。着払いでは受け取りできませんので、あらかじめご注意ください。 |
| 3.3 |
サンプル送付の際は、受託解析サービスQCシートおよび免責事項同意書を同封してください。これらが同封されていない、または記入漏れがある場合、解析サービスに着手できませんので、ご注意ください。 |
| 3.4 |
サンプルを凍結状態のまま輸送する場合は、十分量のドライアイスを同梱の上、冷凍宅急便にてご送付ください。また、解析に必要な量を確保できなかった場合、サンプルの再送をお願いする場合があります。なお、再度サンプルをお送り頂く場合、提携先への再送料金をご負担頂きます。 |
| 3.5 |
サンプル喪失等に対する補償・保険制度はありませんので、貴重なサンプルをご提供頂く際はご注意ください。 |
| 3.6 |
お預かりできるサンプルは、BSL2(バイオセーフティーレベル2)までのものに限られます。感染性が著しく高いサンプル(HIV、HCV、HBVウイルス)に感染していることが確認されている患者由来の検体など)は、お預かりできませんので、予めご了承ください。ヒト臨床サンプルの場合、インフォームドコンセントを得てからご提供ください。また、動物検疫・国際条約等に該当するサンプルの場合、輸送手続きに日数を要する、又はお取り扱いできないことがあります。こうしたサンプルは、弊社提携先にて受入れが不能、又は拒否されたりする場合があります。 |
| 3.7 |
ご提供頂いたサンプルの返却は致しておりません。(サンプルは解析終了時に破棄されます。) |
| 3.8 |
本確認事項を満たさない事で別途費用が発生した場合、お客様に費用のご負担をお願いすることがあります。 |
| 3.9 |
サンプル・業務等から生じた知的財産権・工業所有権・安全性・インフォームドコンセント等の問題については、弊社は一切の責任を負わないものとします。 |
|
| 5. |
キャンセルに関して
本サービスは、海外での解析という性質上、キャンセルをお引き受けできません。やむを得ない理由でキャンセルされる場合、それまでの工程に応じた料金を請求いたします。 |
| 6. |
納品物に関して
納品物は、代理店経由でのお取引の場合、基本的に代理店を介してお送りします。納品データのバックアップは、必ずお客様にてお取りください。弊社でのデータ保管期間は、発送日から90日間です。弊社での保管期間を経過したデータは破棄されます。 |
 価格・納期 価格・納期

|
【価格】
| サービス名 |
税別価格 |
型番 |
空間トランスクリプトームシークエンス(10x Genomics Visium)
受託解析サービス |
お問合せ |
F-BMK-Visium-[サンプル数] |
※本サービスは、海外業務提携先で実施する解析のため、上記の解析料金の他、サンプル海外輸送費が別途発生します。
【納期】
サンプル受領後約 2.5~3 か月
※サンプルQC合格の場合 |
※本サイトの情報は、Biomarker Technologies(BMKGene)社のホームページに掲載されている情報や画像を一部引用しています。
|